Identifizierung von Coronavirus-Virulenzfaktoren für die Entwicklung von RNA-Impfstoffen mit Twist Oligo-Pools
Die Welt spürt weiterhin die Auswirkungen der globalen Pandemie, die durch SARS-CoV-2 verursacht wird, das Virus, das zu COVID-19 führt. Die Pandemie hat zweifellos das Leben auf der Erde verändert. Aber während wir weiter kämpfen, geben uns Anzeichen für Fortschritt neue Hoffnung. Die Forschungsgemeinschaft ist besonders betroffen. Wissenschaftler haben sich schnell mobilisiert, um verschiedene Fragen zu beantworten: wie verursacht dieses Virus eine Infektion, wie wird es übertragen und wie mutiert es? Eine solche, hier vorgestellte Gruppe zeigt, wie Twist Oligo-Pools verwendet werden können, um aufregende Innovationen voranzutreiben und uns zu einem besseren Verständnis dessen verhelfen, wie SARS-CoV-2 unser Immunsystem kapert.
To cause infection, COVID-19 must enter a host cell and release its RNA genome. Virulence is established via replication of this genome, and production of new viral particles through the host cell’s expression of viral genes (Boopathi et al., 2020). Our cells have a number of innate mechanisms that restrict viral replication including RNA degrading proteins and genetic checkpoints alerting the immune system that “this cell has been infected.” These systems work in tandem with the adaptive immune system to halt virulence.
Im Wettlauf ums Überleben haben einige Viren jedoch eine Resistenz gegen unser angeborenes Immunsystem entwickelt. Bei einigen RNA-Viren bietet die Stabilität ihres Genoms Schutz vor RNA-abbauenden Enzymen. Die Stabilität von RNA wird erhöht, wenn sie durch Basenpaarung mit sich selbst komplexe Sekundärstrukturen bildet. Diese Sekundärstrukturen können für die antivirale Aktivität der Wirtszelle unempfindlich sein oder diese sogar aktiv blockieren, sodass sich das Virus unbemerkt replizieren kann.
Therapeutika, die auf die Stabilität sich schnell entwickelnder viraler RNA abzielen, können es dem Immunsystem des Wirts ermöglichen, die Virusinfektion effektiv abzubauen und zu beseitigen. In einem kürzlich veröffentlichten Artikel in Biochemical and Biophysical Research Communications beschreiben Wakida et al. die Entwicklung einer Methode, mit der RNA-Sequenzen, die von RNA- und DNA-Viren stammen und zur RNA-Stabilität in Wirtszellen beitragen, expansiv interrogiert werden. Der leitende Autor, Professor Nobuyoshi Akimitsu, beschreibt diese Studie als einen Schritt zur Verbesserung der RNA-Medizin.
„mRNA ist instabil und die Translation in der Zelle ist oft schlecht. Durch die Untersuchung natürlicher Elemente, die die Stabilität und Struktur von mRNA verbessern, hoffen wir, die Funktionalität von mRNA als Arzneimittel zu verbessern“, erklärte Prof. Akimitsu.
Die in dieser Studie entwickelte Methode wird als „Fate-seq“ bezeichnet. Fate-seq arbeitet nach dem Prinzip, dass stabile RNA-Sequenzen in einer Säugetierzelle viel langsamer abgebaut werden als instabile Sequenzen. Durch Binden von zu beobachtenden RNA-Sequenzen an ein Signalmolekül wie grün fluoreszierendes Protein und Einbringen in Zellen kann der Abbau direkt gemessen werden. Zellen, die im Laufe der Zeit das stärkste Fluoreszenzsignal zeigen, enthalten Reporter, die an die stabilsten RNA-Strukturen gebunden sind. Die Sequenzierung von Zellen mit dem stärksten Signal ermöglicht dann das präzise Mapping hochstabiler RNA-Sequenzen.
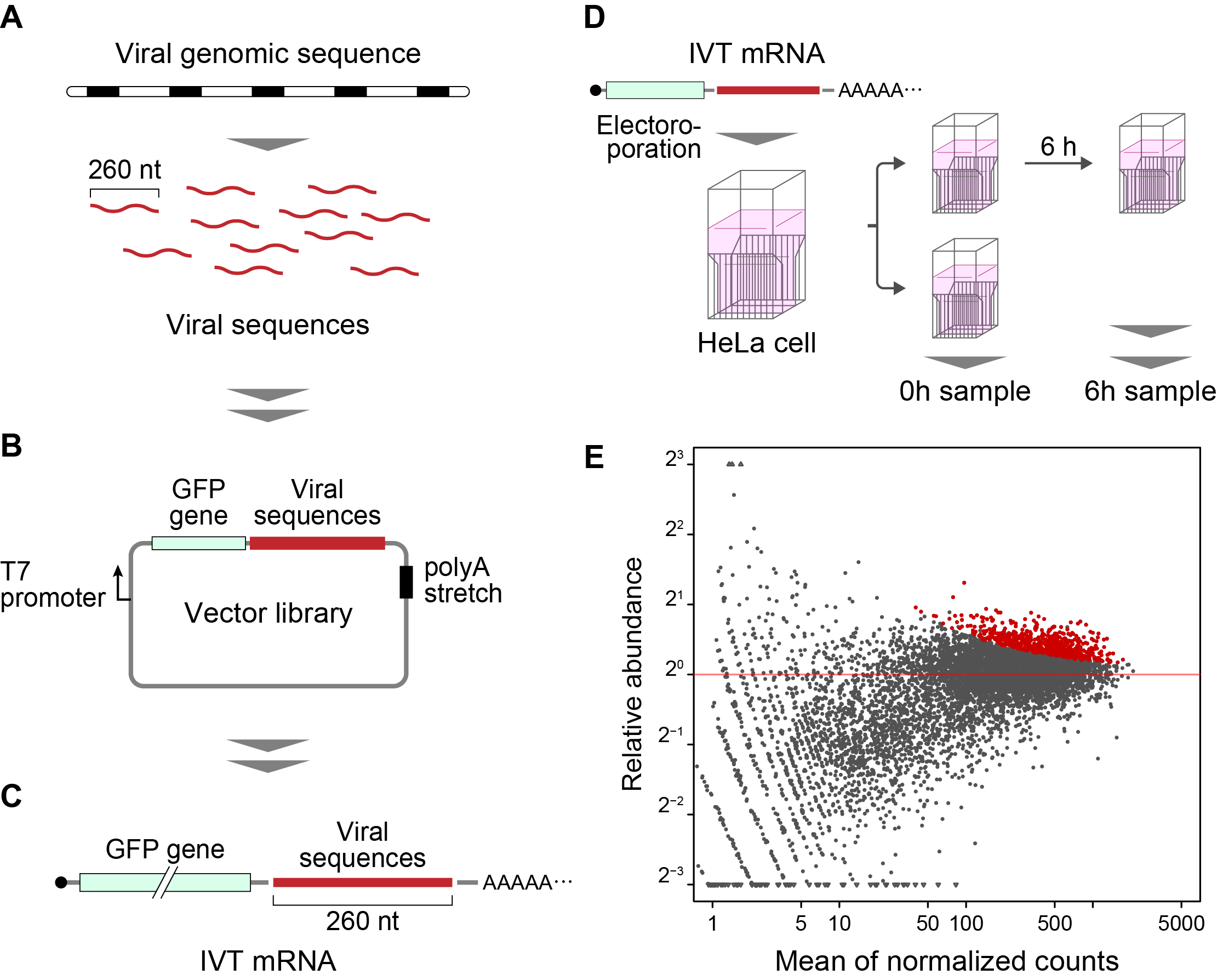
Wakida et al. verwendeten Twist Bioscience Oligo-Pools, um das Genom von 26 verschiedenen Viren zu scannen. Jedes Virusgenom wurde in Abschnitte von 260 Nukleotiden (nt) mit 200 nt Überlappung unterteilt. Prof. Akimitsu betonte die Bedeutung der Verwendung hochwertiger Oligonukleotide in der Studie.
“When doing a genome scan like Fate-seq, you need to be sure you are covering as much of the genome as possible, as evenly as possible. Because of the good uniformity and low error of Twist Oligo Pools, we can get even coverage across the viral genomes, and know we do not miss any potentially critical data.” Twist Oligo Pools can be synthesized up to 300 nucleotides, with an industry leading error rate of 1:2000.
In der Studie deckte Fate-seq zwei hochkonservierte RNA-stabilisierende Regionen (COV001 und COV002) im schweren Coronavirus (SARS-CoV) und SARS-CoV-2 auf. Ergebnisse von Wakida et al. stellten eine Änderung der Sekundärstruktur von COV001 in SARS-CoV-2 fest, die für SARS-CoV nicht vermutet wurde. Die Autoren interpretieren, dass „dieser potenzielle Unterschied in der Sekundärstruktur zur Virulenz von SARS-CoV-2 beitragen kann. Die identifizierten Sequenzen, die die Stabilität der Virus-RNA und die Bindung der RNA-bindenden Proteine an diese Sequenzen beeinflussen könnten, könnten mögliche Ziele für die Behandlung von viralen Infektionskrankheiten einschließlich COVID-19 darstellen.“
Development of COVID-19 vaccines are currently in high-demand, many consisting of messenger RNA to activate the host immune system (Callaway, 2020). New technologies like Fate-seq to identify stabilizing molecules will offer significant benefits to the RNA vaccine development field, allowing more stable and efficacious vaccines to be produced. Moreover, Prof. Akimitsu reiterated “RNA stability is important for the viral life cycle. In theory, by inhibiting stability, we can reduce viral proliferation and virulence.” Fate-seq is therefore an expeditious and promising approach to spotlight critical genomic regions for further characterization as targets for antiviral therapeutics.
Titelbild von Fusion Medical Animation auf Unsplash
Was denken Sie?
Gefällt mir
Gefällt mir nicht
Gefällt mir sehr
Überraschend
Interessant
