Performance quelle que soit l’échelle
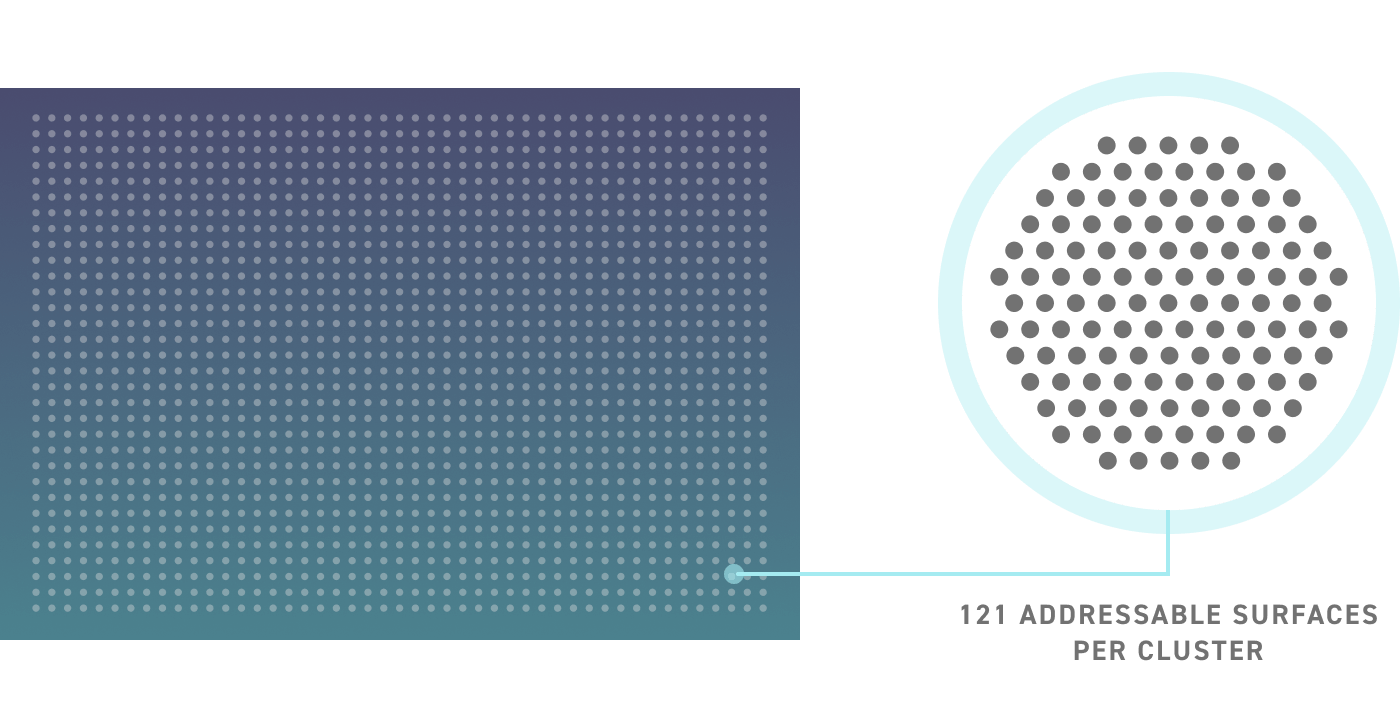
Notre plateforme de synthèse d’ADN révolutionnaire, basée sur le silicone, est capable de produire plus d’un million d’oligos d’ADNss uniques en une seule fois. Chaque puce contient des milliers de clusters distincts, chacun contenant 121 surfaces adressables individuellement capables de synthétiser une séquence d’oligonucléotide unique. Une représentation de la technologie des puces en silicone se trouve ci-dessous.
En raison de l’ampleur de la capacité de synthèse d’ADN sur la puce, il n’y a pratiquement aucune limite au nombre d’oligonucléotides uniques qui peuvent être assemblés dans un pool. Ne limitez pas le résultat de votre application, commandez des pools d’oligonucléotides spécifiquement adaptés à votre expérience.
Plus d’espace pour l’innovation
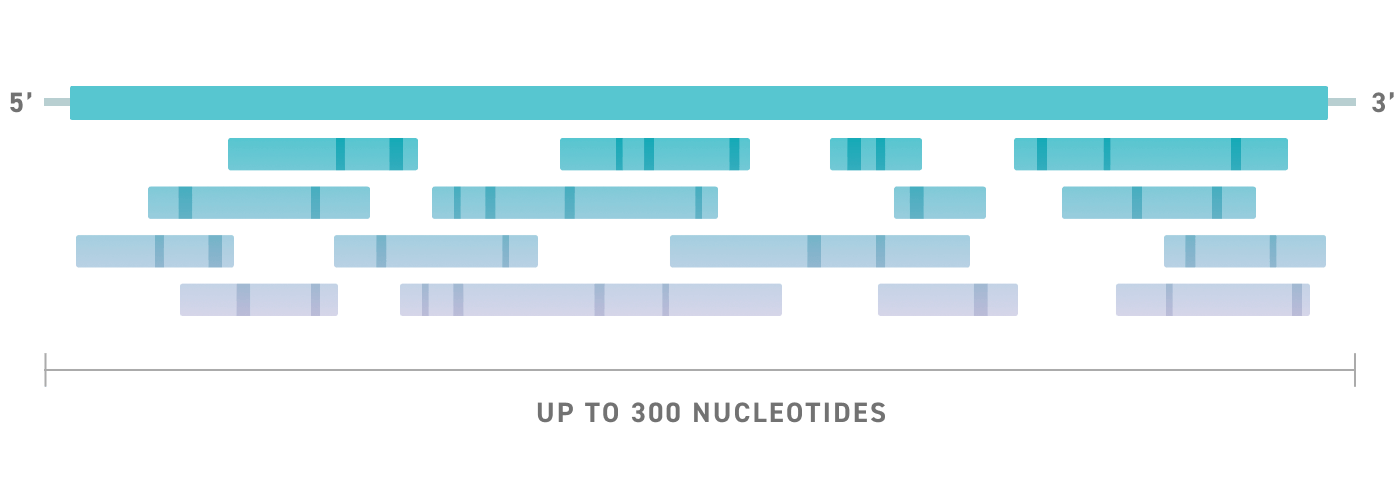
Ne soyez pas limité par la longueur. Encodez les éléments et les séquences dont vous avez besoin, y compris les promoteurs, les exhausteurs, les guides et les sondes, le tout dans une architecture étendue. Avec des longueurs d’oligo allant jusqu’à 300 nt, il est possible de coder plusieurs éléments dans une seule structure, ce qui vous permet d’en faire plus avec chaque oligonucléotide.
Criblage réussi en une seule fois
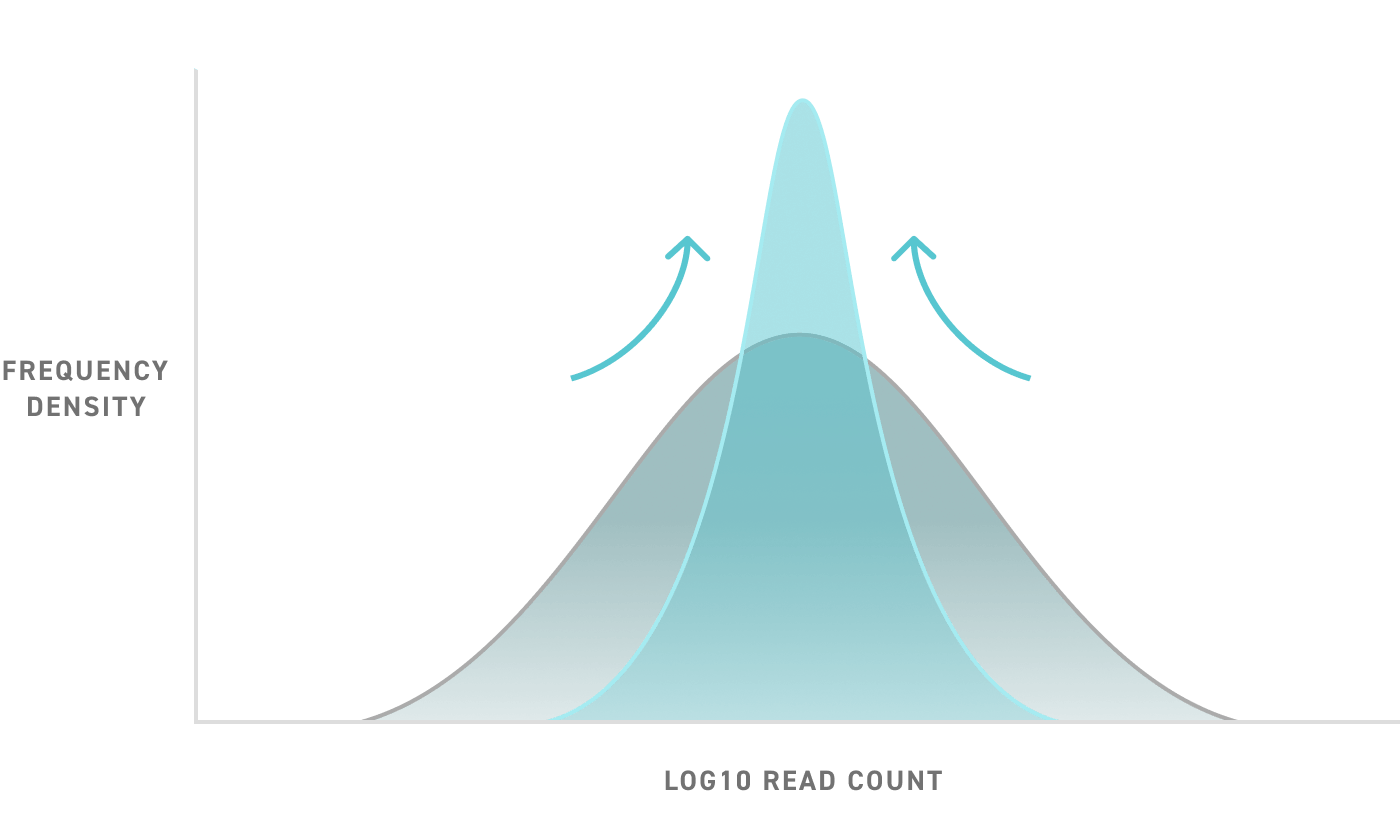
L’uniformité inégalée du pool sans biais permet d’obtenir plus de cibles uniques par crible. Cela signifie que moins d’efforts sont nécessaires pour obtenir la couverture de données dont vous avez besoin, ce qui vous fait gagner du temps et de l’argent.
Soyez sûr(e) de vos données
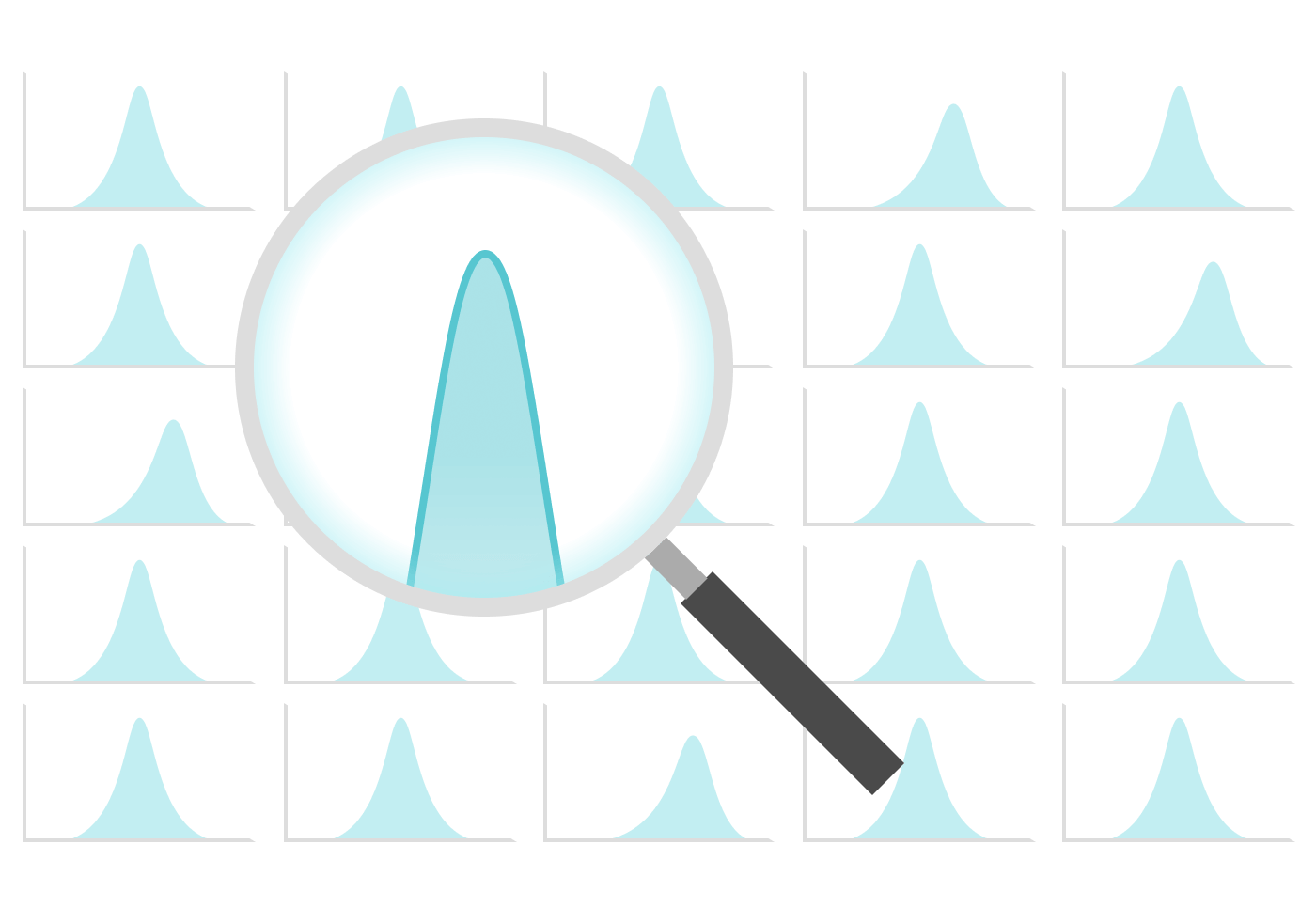
Grâce à la précision et à l’uniformité des pools d’oligonucléotides Twist Bioscience, vous pouvez être sûr(e) de la qualité de vos résultats expérimentaux. Ne perdez pas un seul élément de données en raison d’erreurs de synthèse ou du manque d’efficacité. Assurez la meilleure représentation des oligonucléotides et un faible taux d’erreur pour les oligonucléotides de toute longueur jusqu’à 300 nt, et les pools de toute taille. Optimisez ces pools d’oligo de qualité exceptionnelle à l’échelle et à la longueur précises requises pour votre application.
Dites-nous ce sur quoi vous souhaitez obtenir plus d’informations. Nous sommes là pour vous aider !
Nous contacter
Performance quelle que soit l’échelle
Notre plateforme de synthèse d’ADN révolutionnaire, basée sur le silicone, est capable de produire plus d’un million d’oligos d’ADNss uniques en une seule fois. Chaque puce contient des milliers de clusters distincts, chacun contenant 121 surfaces adressables individuellement capables de synthétiser une séquence d’oligonucléotide unique. Une représentation de la technologie des puces en silicone se trouve ci-dessous.
En raison de l’ampleur de la capacité de synthèse d’ADN sur la puce, il n’y a pratiquement aucune limite au nombre d’oligonucléotides uniques qui peuvent être assemblés dans un pool. Ne limitez pas le résultat de votre application, commandez des pools d’oligonucléotides spécifiquement adaptés à votre expérience.

Plus d’espace pour l’innovation
Ne soyez pas limité par la longueur. Encodez les éléments et les séquences dont vous avez besoin, y compris les promoteurs, les exhausteurs, les guides et les sondes, le tout dans une architecture étendue. Avec des longueurs d’oligo allant jusqu’à 300 nt, il est possible de coder plusieurs éléments dans une seule structure, ce qui vous permet d’en faire plus avec chaque oligonucléotide.
Criblage réussi en une seule fois
L’uniformité inégalée du pool sans biais permet d’obtenir plus de cibles uniques par crible. Cela signifie que moins d’efforts sont nécessaires pour obtenir la couverture de données dont vous avez besoin, ce qui vous fait gagner du temps et de l’argent.

Soyez sûr(e) de vos données
Grâce à la précision et à l’uniformité des pools d’oligonucléotides Twist Bioscience, vous pouvez être sûr(e) de la qualité de vos résultats expérimentaux. Ne perdez pas un seul élément de données en raison d’erreurs de synthèse ou du manque d’efficacité. Assurez la meilleure représentation des oligonucléotides et un faible taux d’erreur pour les oligonucléotides de toute longueur jusqu’à 300 nt, et les pools de toute taille. Optimisez ces pools d’oligo de qualité exceptionnelle à l’échelle et à la longueur précises requises pour votre application.

Dites-nous ce sur quoi vous souhaitez obtenir plus d’informations. Nous sommes là pour vous aider !
Nous contacter
Pools d’oligonucléotides
Les Twist Oligo Pools sont des ensembles très divers d’oligonucléotides à simple brin synthétisés à l’aide de notre technologie d’écriture de l’ADN sur silicone. Notre plateforme de synthèse permet la production massivement parallèle de centaines de milliers d’oligonucléotides précis et de haute qualité par cycle, ce qui permet de générer des pools d’oligonucléotides complexes et diversifiés pour des applications telles que le criblage CRISPR.
Comment cela fonctionne-t-il ?
Vous nous fournissez vos séquences d’oligonucléotides et nous synthétisons les banques de pools d’oligonucléotides conçus par l’utilisateur, ce qui vous permet de consacrer votre temps à l’expérimentation et à la découverte.
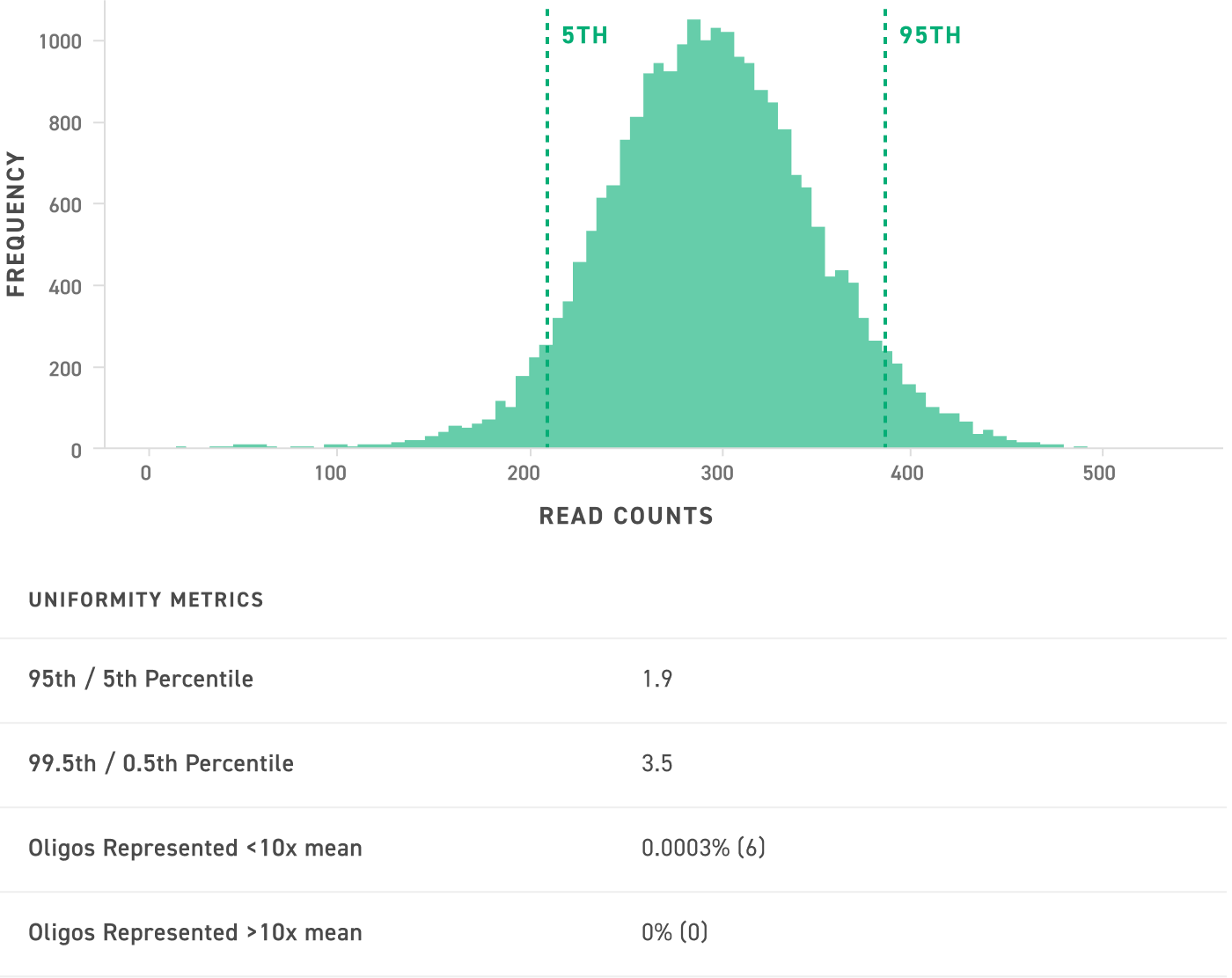
Synthèse d’oligonucléotides extrêmement uniforme
Les données de contrôle de qualité NGS provenant d’un pool d’oligonucléotides standard contenant 23 000 oligonucléotides 90-mer montrent l’uniformité du pool à une couverture de lecture de 300x. Le tableau correspondant indique les paramètres d’uniformité pour ce pool. Les oligonucléotides Twist Bioscience sont synthétisés sans biais avec une grande uniformité et une représentation complète des oligonucléotides.
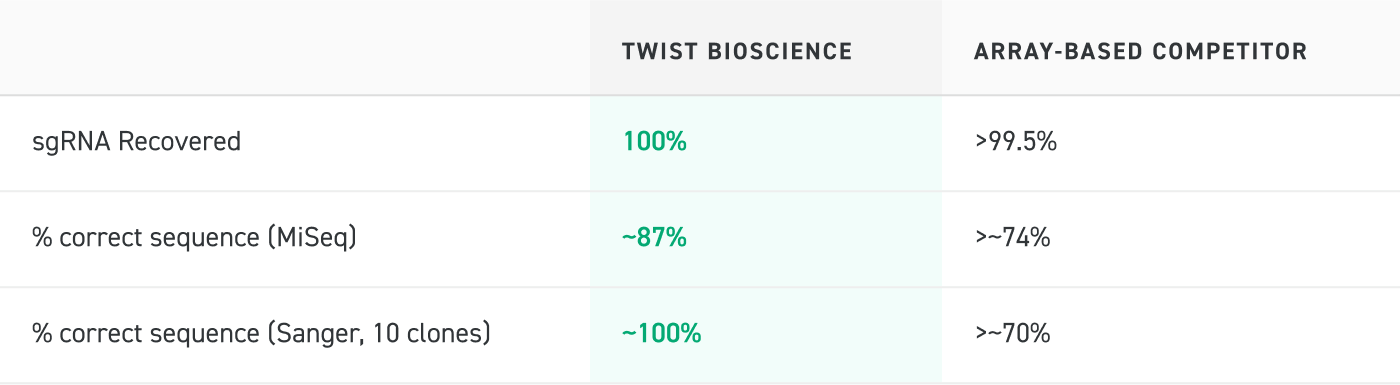
Analyse de séquençage des pool d’oligonucléotides
L’analyse de séquençage des pools d’oligonucléotides générés par Twist Bioscience et d’un concurrent de type matrice démontre que les pools d’oligonucléotide Twist Bioscience contiennent 100 % des séquences escomptées et un pourcentage plus élevé de séquences correctes que le pool du concurrent.
Spécifications
| Longueur oligonucléotide | Jusqu’à 300 nt |
| Taille du pool d'oligonucléotides | Aucune limite sur la taille du pool d’oligonucléotides |
| Rendement | >0,2 fmol moyen de chaque oligonucléotide |
| Uniformité | >90 % oligonucléotides représentés dans un écart < 2,0x de la moyenne |
| Taux d’erreur | Jusqu’à 1 : 3000 |
| Délais d’exécution | En seulement 3 jours* |
| Prix | Prix parmi les meilleurs du secteur |
*Le délai d'exécution pour l'offre standard de pools d'oligonucléotides varie en fonction de la longueur et de la complexité de la séquence. Les pools d'oligonucléotides d'une longueur de 20 à 121 nucléotides varient entre 3 à 6 jours ouvrables, ceux de 121 à 200 nucléotides varient entre 3 à 8 jours ouvrables, et ceux de 201 à 300 nucléotides varient entre 5 à 10 jours ouvrables.
Dites-nous ce sur quoi vous souhaitez obtenir plus d’informations. Nous sommes là pour vous aider !
Nous contacter
Pools d’oligonucléotides
Les Twist Oligo Pools sont des ensembles très divers d’oligonucléotides à simple brin synthétisés à l’aide de notre technologie d’écriture de l’ADN sur silicone. Notre plateforme de synthèse permet la production massivement parallèle de centaines de milliers d’oligonucléotides précis et de haute qualité par cycle, ce qui permet de générer des pools d’oligonucléotides complexes et diversifiés pour des applications telles que le criblage CRISPR.
Comment cela fonctionne-t-il ?
Vous nous fournissez vos séquences d’oligonucléotides et nous synthétisons les banques de pools d’oligonucléotides conçus par l’utilisateur, ce qui vous permet de consacrer votre temps à l’expérimentation et à la découverte.

Synthèse d’oligonucléotides extrêmement uniforme
Les données de contrôle de qualité NGS provenant d’un pool d’oligonucléotides standard contenant 23 000 oligonucléotides 90-mer montrent l’uniformité du pool à une couverture de lecture de 300x. Le tableau correspondant indique les paramètres d’uniformité pour ce pool. Les oligonucléotides Twist Bioscience sont synthétisés sans biais avec une grande uniformité et une représentation complète des oligonucléotides.

Analyse de séquençage des pool d’oligonucléotides
L’analyse de séquençage des pools d’oligonucléotides générés par Twist Bioscience et d’un concurrent de type matrice démontre que les pools d’oligonucléotide Twist Bioscience contiennent 100 % des séquences escomptées et un pourcentage plus élevé de séquences correctes que le pool du concurrent.

Spécifications
| Longueur oligonucléotide | Jusqu’à 300 nt |
| Taille du pool d'oligonucléotides | Aucune limite sur la taille du pool d’oligonucléotides |
| Rendement | >0,2 fmol moyen de chaque oligonucléotide |
| Uniformité | >90 % oligonucléotides représentés dans un écart < 2,0x de la moyenne |
| Taux d’erreur | Jusqu’à 1 : 3000 |
| Délais d’exécution | En seulement 3 jours* |
| Prix | Prix parmi les meilleurs du secteur |
*Le délai d'exécution pour l'offre standard de pools d'oligonucléotides varie en fonction de la longueur et de la complexité de la séquence. Les pools d'oligonucléotides d'une longueur de 20 à 121 nucléotides varient entre 3 à 6 jours ouvrables, ceux de 121 à 200 nucléotides varient entre 3 à 8 jours ouvrables, et ceux de 201 à 300 nucléotides varient entre 5 à 10 jours ouvrables.
Dites-nous ce sur quoi vous souhaitez obtenir plus d’informations. Nous sommes là pour vous aider !
Nous contacter
Nouveaux pools d’oligonucléotides clonés
La qualité des pools d’oligonucléotides est la base d’une expérience réussie. Les erreurs de synthèse ou de clonage peuvent facilement fausser la qualité des pools d’oligonucléotides, entraînant une sur- ou une sous-représentation des séquences souhaitées. Twist propose désormais un service de clonage optimisé qui vous permet d’éviter davantage les difficultés liées à l’essai des conditions d’amplification de la PCR, à la sélection des polymérases et des paires d’amorces appropriées, ainsi qu’à la conception d’un flux de travail de clonage. Évitez les problèmes et laissez Twist synthétiser et cloner des pools d’oligonucléotides pour vous.
Comment cela fonctionne-t-il ?
À deux pas de vos pools d'oligonucléotides clonés personnalisés. Vous nous fournissez vos séquences d’oligonucléotides et nous synthétisons, amplifions et clonons les banques de pools d’oligonucléotides conçus par l’utilisateur, ce qui vous permet de consacrer votre temps à l’expérimentation et à la découverte.
Les pools d'oligonucléotides clonés de Twist Bioscience, quelle que soit leur longueur, présentent une grande uniformité et un faible taux d'erreur
Données de performance des pools d'oligonucléotides amplifiés et clonés d'une longueur inférieure à 150 nucléotides, même avec un contenu GC élevé :
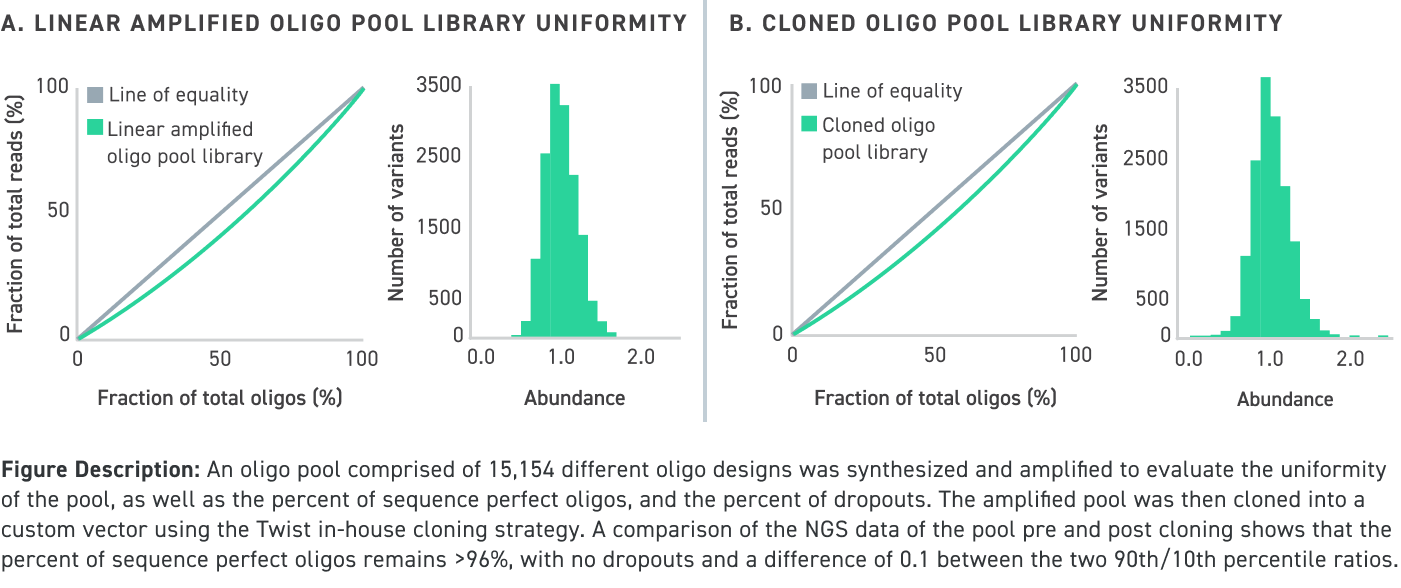
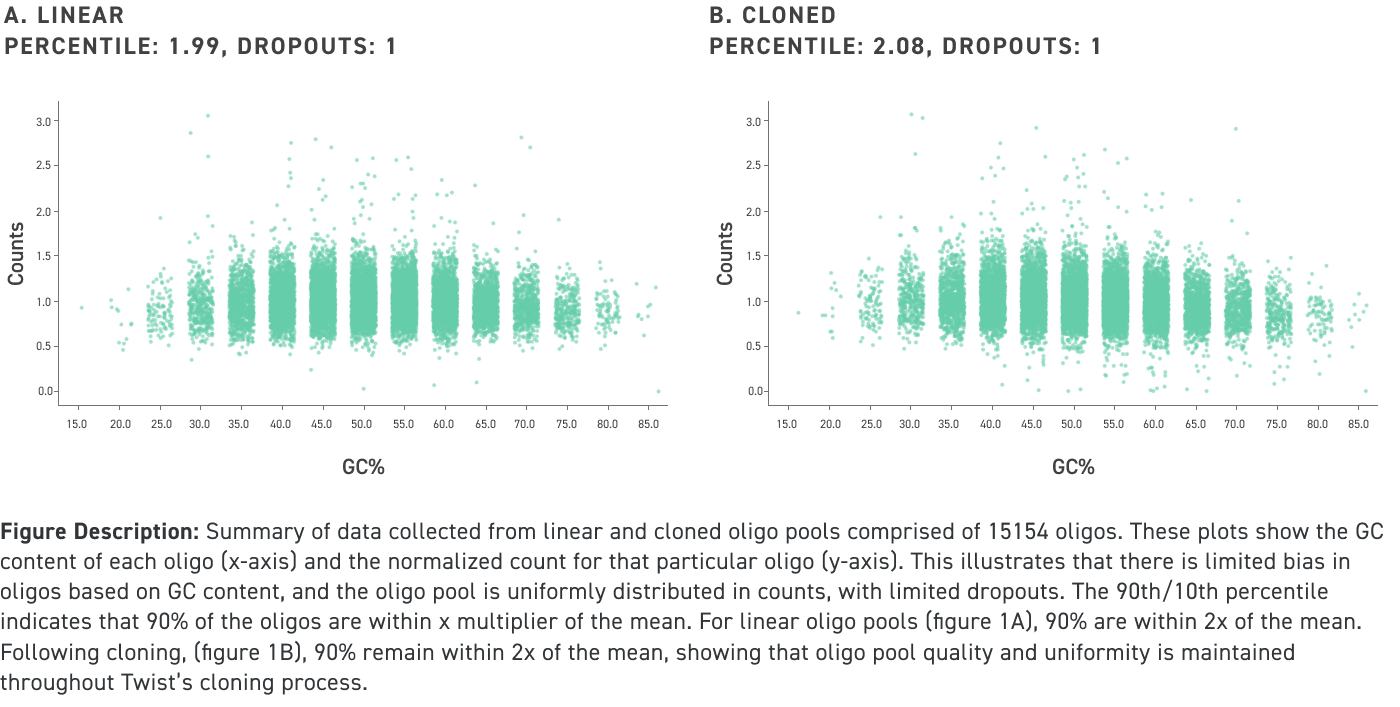
Données de performance des pools d'oligonucléotides amplifiés et clonés d'une longueur allant jusqu’à 300 nucléotides, même avec un contenu GC élevé :
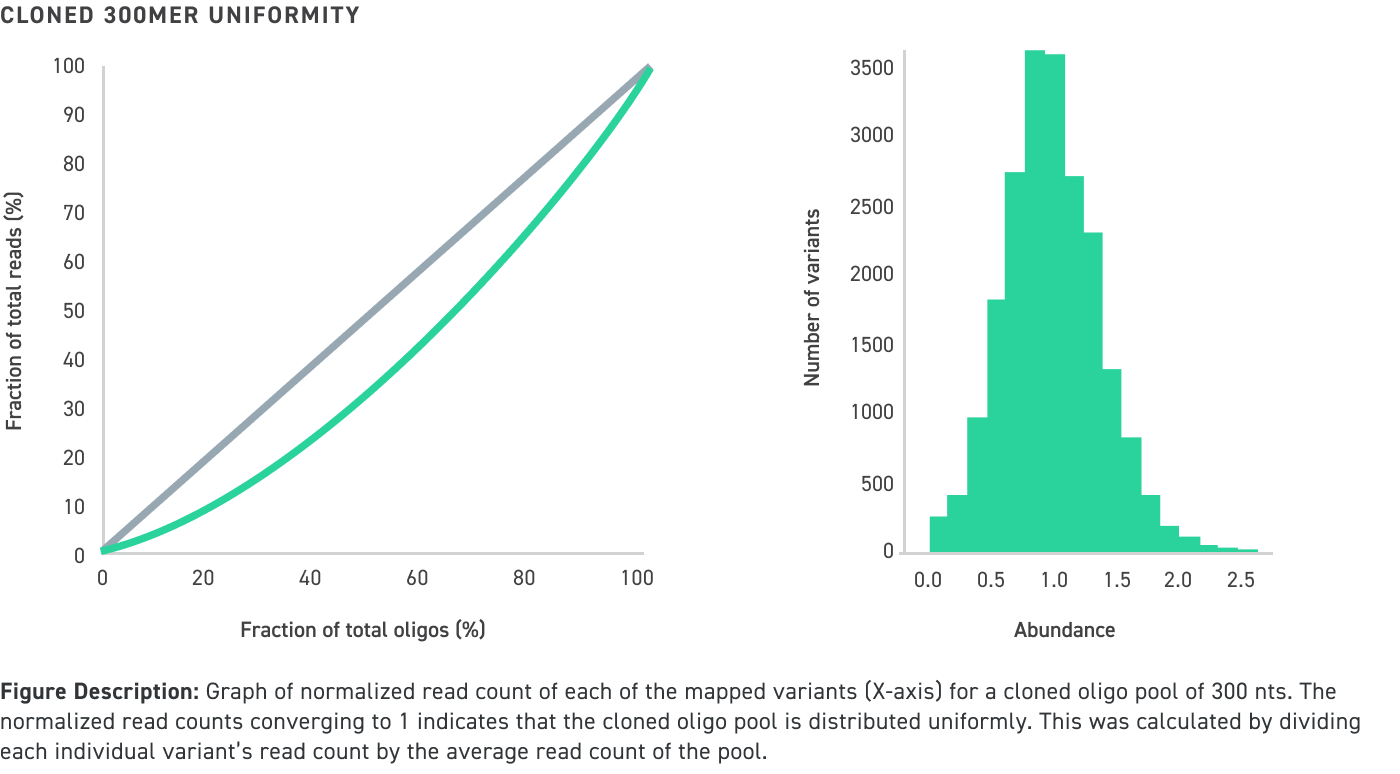
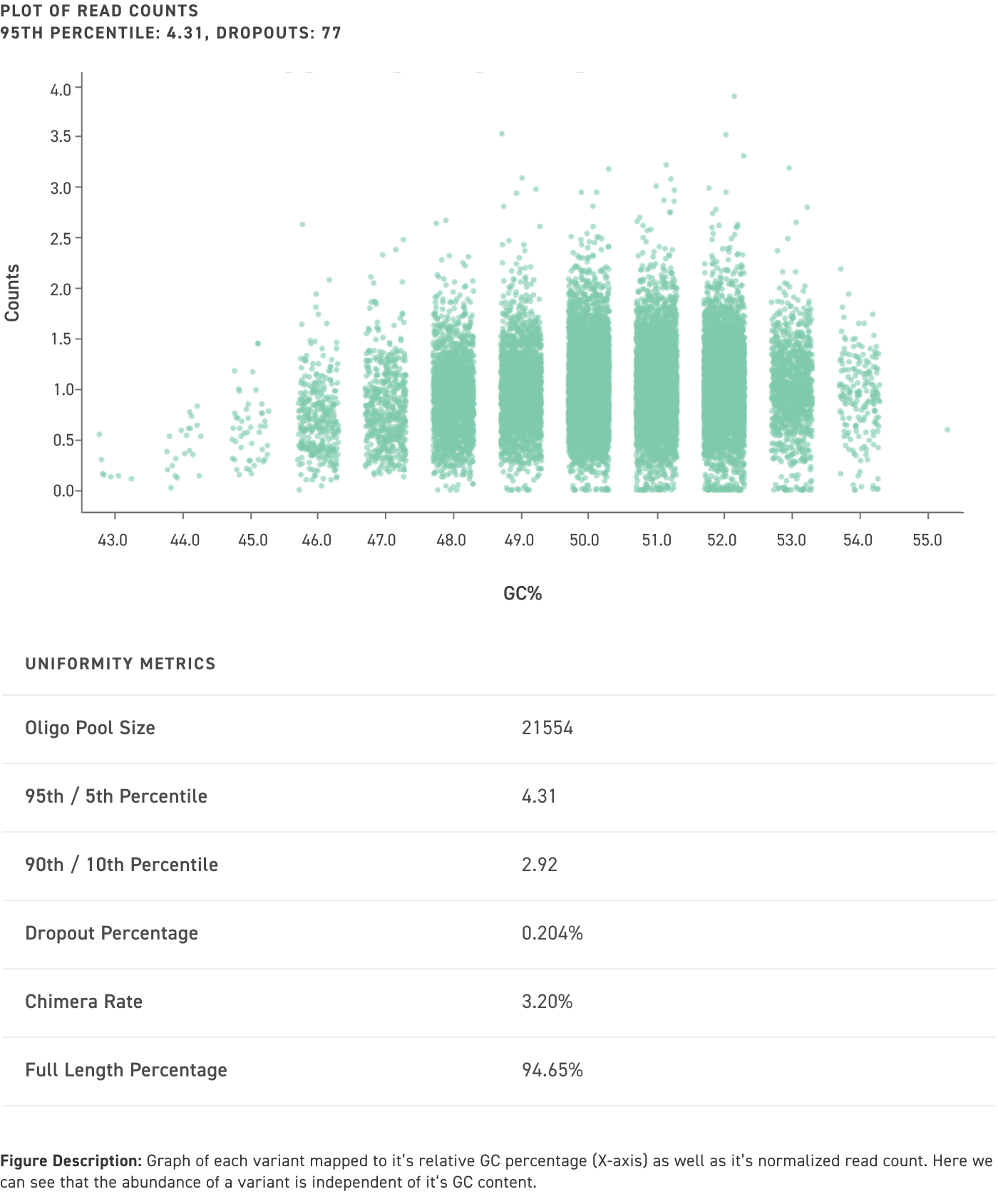
Obtenez une qualité et une précision optimales de vos données expérimentales
Les pools d'oligonucléotides clonés de Twist présentent non seulement un faible taux d'erreur et une grande uniformité, mais aussi un faible taux de chimères. Les chimères sont des molécules hybrides indésirables résultant de conditions de PCR sous-optimales qui mènent à une amplification et à une recombinaison incorrectes de différents oligonucléotides au sein du pool d'oligonucléotides. Consultez notre illustration ci-dessous pour un exemple d'un pool cloné sur la cible par rapport à un pool cloné hors cible qui contient des chimères, ce qui mène finalement à des pools d'oligonucléotides clonés qui sont composés de séquences non désirées :
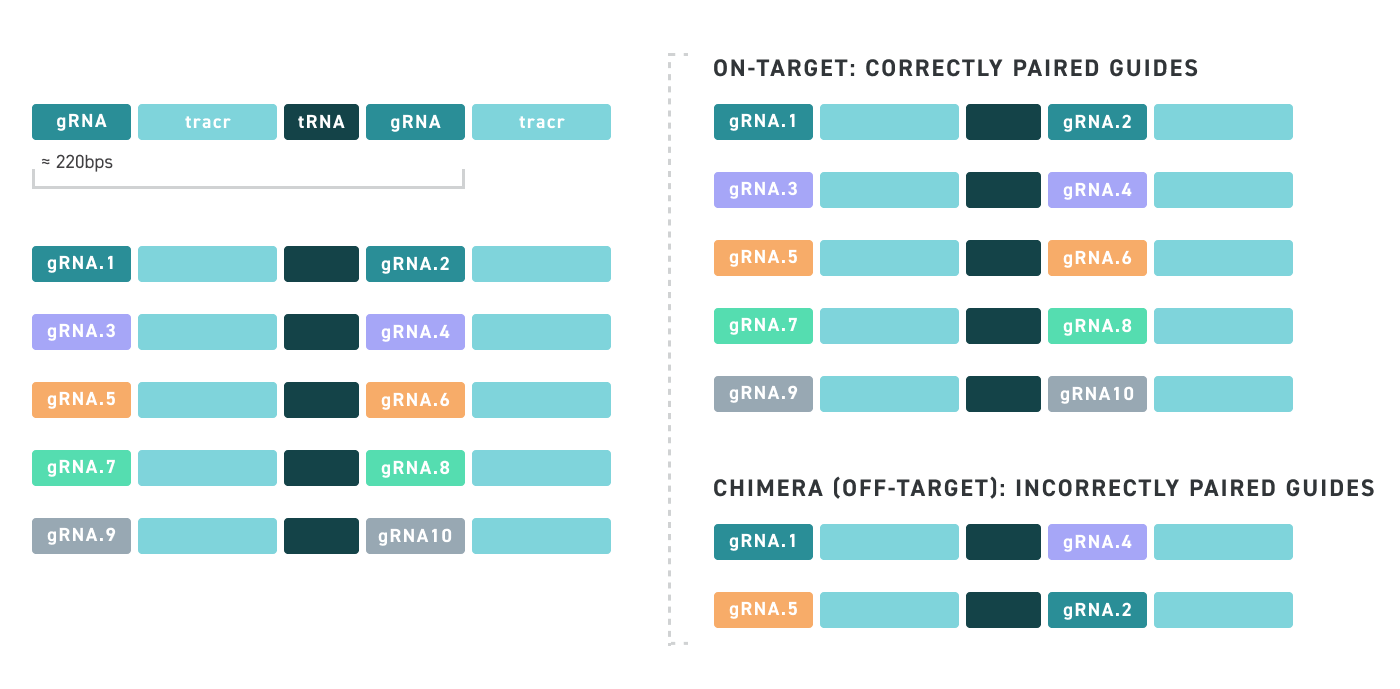
Avec Twist, vous n'avez plus à vous soucier des chimères ! Vérifiez par vous-même :
Amplification traditionnelle vs performance de la méthode d'amplification Twist en interne
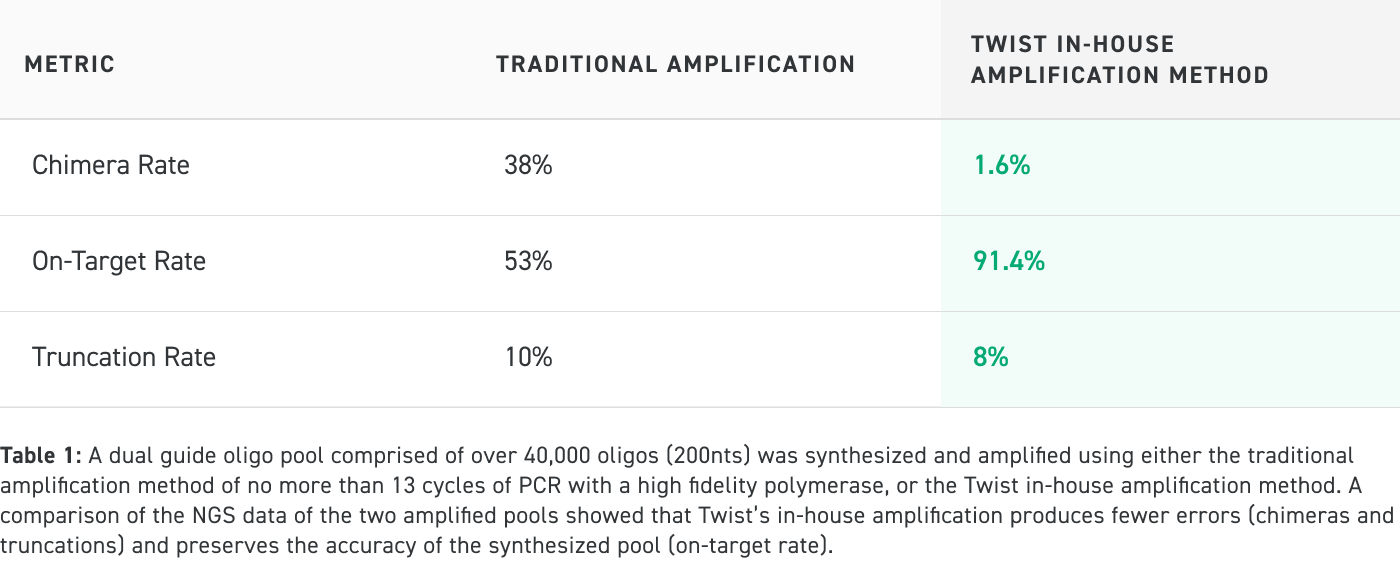
Spécifications
| Longueur des oligonucléotides Jusqu'à 300 nt |
| Taille du pool d'oligonucléotides Pas de limite de taille |
| Rendement >0,2 fmol moyenne de chaque oligonucléotide |
| Uniformité >90 % oligonucléotides représentés dans une fourchette < 5,0x de la moyenne |
| Taux de chimères Minimal 1,5%* |
| Délai d'exécution Pas plus de 4 semaines* |
*Conditions générales : Le délai d'exécution de base pour les pool d'oligonucléotides clonés, qui comprend le clonage et l'amplification, jusqu'à 100 et 300 nucléotides est de 4 à 6 semaines et 6 à 8 semaines, respectivement. Un délai supplémentaire de 2 semaines sera ajouté à ce délai de base pour tous les pool d'oligonucléotides clonés, quelle que soit leur longueur, qui nécessitent l'intégration d'un nouveau vecteur. Le taux de chimères, le taux de pertes et l'uniformité varient en fonction de la complexité de la séquence.
Dites-nous ce sur quoi vous souhaitez obtenir plus d’informations. Nous sommes là pour vous aider !
Nous contacter
Nouveaux pools d’oligonucléotides clonés
La qualité des pools d’oligonucléotides est la base d’une expérience réussie. Les erreurs de synthèse ou de clonage peuvent facilement fausser la qualité des pools d’oligonucléotides, entraînant une sur- ou une sous-représentation des séquences souhaitées. Twist propose désormais un service de clonage optimisé qui vous permet d’éviter davantage les difficultés liées à l’essai des conditions d’amplification de la PCR, à la sélection des polymérases et des paires d’amorces appropriées, ainsi qu’à la conception d’un flux de travail de clonage. Évitez les problèmes et laissez Twist synthétiser et cloner des pools d’oligonucléotides pour vous.
Comment cela fonctionne-t-il ?
À deux pas de vos pools d'oligonucléotides clonés personnalisés. Vous nous fournissez vos séquences d’oligonucléotides et nous synthétisons, amplifions et clonons les banques de pools d’oligonucléotides conçus par l’utilisateur, ce qui vous permet de consacrer votre temps à l’expérimentation et à la découverte.

Les pools d'oligonucléotides clonés de Twist Bioscience, quelle que soit leur longueur, présentent une grande uniformité et un faible taux d'erreur
Données de performance des pools d'oligonucléotides amplifiés et clonés d'une longueur inférieure à 150 nucléotides, même avec un contenu GC élevé :


Données de performance des pools d'oligonucléotides amplifiés et clonés d'une longueur allant jusqu’à 300 nucléotides, même avec un contenu GC élevé :


Obtenez une qualité et une précision optimales de vos données expérimentales
Les pools d'oligonucléotides clonés de Twist présentent non seulement un faible taux d'erreur et une grande uniformité, mais aussi un faible taux de chimères. Les chimères sont des molécules hybrides indésirables résultant de conditions de PCR sous-optimales qui mènent à une amplification et à une recombinaison incorrectes de différents oligonucléotides au sein du pool d'oligonucléotides. Consultez notre illustration ci-dessous pour un exemple d'un pool cloné sur la cible par rapport à un pool cloné hors cible qui contient des chimères, ce qui mène finalement à des pools d'oligonucléotides clonés qui sont composés de séquences non désirées :

Avec Twist, vous n'avez plus à vous soucier des chimères ! Vérifiez par vous-même :
Amplification traditionnelle vs performance de la méthode d'amplification Twist en interne

Spécifications
| Longueur des oligonucléotides Jusqu'à 300 nt |
| Taille du pool d'oligonucléotides Pas de limite de taille |
| Rendement >0,2 fmol moyenne de chaque oligonucléotide |
| Uniformité >90 % oligonucléotides représentés dans une fourchette < 5,0x de la moyenne |
| Taux de chimères Minimal 1,5%* |
| Délai d'exécution Pas plus de 4 semaines* |
*Conditions générales : Le délai d'exécution de base pour les pool d'oligonucléotides clonés, qui comprend le clonage et l'amplification, jusqu'à 100 et 300 nucléotides est de 4 à 6 semaines et 6 à 8 semaines, respectivement. Un délai supplémentaire de 2 semaines sera ajouté à ce délai de base pour tous les pool d'oligonucléotides clonés, quelle que soit leur longueur, qui nécessitent l'intégration d'un nouveau vecteur. Le taux de chimères, le taux de pertes et l'uniformité varient en fonction de la complexité de la séquence.
Dites-nous ce sur quoi vous souhaitez obtenir plus d’informations. Nous sommes là pour vous aider !
Nous contacter
Banques d’ARN guides pour les cribles CRISPR
Les cribles CRISPR comportent l’élimination, l’interférence ou la modulation précises et à haut débit de l’activité des gènes, ce qui permet de révéler des interactions génotype-phénotype complexes. Les pool d’oligonucléotides sont conçus pour coder plusieurs molécules d’ARN guide par gène.
Les avantages du produit sont les suivants :
- Un contrôle total sur la conception de la banque de guides, évitant les guides non désirés, le gaspillage de séquençage et les ratios limités de guides par gène.
- L’association d’enzymes Cas haute fidélité et d’une synthèse très précise pour des cribles haute fidélité avec des effets hors cible minimaux.
- Des pools incroyablement uniformes et sans biais permettent d’obtenir un rapport signal/bruit amplifié pour une détection maximale des résultats, ce qui réduit les coûts en aval.
- Les oligonucléotides allant jusqu’à 300 nt libèrent de l’espace pour des éléments expérimentaux supplémentaires. Il est facile d’ajouter des guides multiples par oligonucléotide, des homologies génomiques ou des promoteurs minimaux.
Regardez notre webinaire : Criblages CRISPR de nouvelle génération basés sur la transcriptomique en cellule unique
Banques de données génomiques relatives aux puces à sonde recouvrantes
Les banques de puces permettent une étude complète de la fonctionnalité génomique. Les pools d’oligonucléotides peuvent coder des jeux ordonnés de peptides, des séquences codant des ARNm ou des fragments de génome.
Les avantages du produit sont les suivants :
- Le « tiling » détermine les régions génomiques d’intérêt. Réduire le bruit expérimental en excluant les séquences indésirables à l’étape de la conception.
- Les séquences complexes, répétitives ou palindromiques qui ne peuvent généralement pas être synthétisées sous forme de gènes peuvent être codées dans des oligonucléotides.
- Les structures peptidiques plus importantes ou les architectures de génomes peuvent être codées dans des oligos de 300 nt de long.
- La synthèse uniforme minimise le suréchantillonnage nécessaire à un criblage complet.
Lisez notre article : Identification des facteurs de virulence du coronavirus pour le développement de vaccins à ARN avec la méthode fate-seq basée sur le pool d’oligonucléotides de Twist Bioscience.
Cribles de contrôle génétique à haut débit
La compréhension des éléments génétiques contrôlant l’expression et la régulation des gènes trouve une large application dans la biologie de synthèse et le développement de traitements potentiels des maladies. Les pool d’oligonucléotides permettent de réaliser des cribles à haut débit du contrôle génétique, notamment le profilage de l’expression génétique et les applications basées sur les rapporteurs.
Les avantages du produit sont les suivants :
- Des pools de haute qualité permettent de vérifier la fonctionnalité de chaque base.
- Une grande uniformité signifie que vous criblerez moins pour trouver vos réponses.
- Des pools de 300 nt vous donnent la possibilité d’ajouter plusieurs éléments ou régions d’homologie.
Regardez notre webinaire : Criblage épitranscriptomique sur TechNetworks
Hybridation in situ en fluorescence
L’hybridation in situ par fluorescence (FISH) permet de visualiser directement l’abondance et l’organisation spatiale des acides nucléiques dans les échantillons biologiques. Les pool d’oligonucléotides peuvent coder des sondes qui, lorsqu’elles sont liées à des marqueurs fluorescents, permettent une visualisation programmable et réglable d’un grand nombre de cibles.
Les avantages du produit sont les suivants :
- Développement rapide de sondes FISH
- Le contrôle de la séquence permet un ciblage efficace
- Des oligonucléotides plus longs permettent un ciblage plus spécifique
Regardez notre webinaire : Production rapide et efficace de sondes FISH à l’aide de pools d’oligonucléotides, avec Nicola Crosetto
Banques d’ARN guides pour les cribles CRISPR
Les cribles CRISPR comportent l’élimination, l’interférence ou la modulation précises et à haut débit de l’activité des gènes, ce qui permet de révéler des interactions génotype-phénotype complexes. Les pool d’oligonucléotides sont conçus pour coder plusieurs molécules d’ARN guide par gène.
Les avantages du produit sont les suivants :
- Un contrôle total sur la conception de la banque de guides, évitant les guides non désirés, le gaspillage de séquençage et les ratios limités de guides par gène.
- L’association d’enzymes Cas haute fidélité et d’une synthèse très précise pour des cribles haute fidélité avec des effets hors cible minimaux.
- Des pools incroyablement uniformes et sans biais permettent d’obtenir un rapport signal/bruit amplifié pour une détection maximale des résultats, ce qui réduit les coûts en aval.
- Les oligonucléotides allant jusqu’à 300 nt libèrent de l’espace pour des éléments expérimentaux supplémentaires. Il est facile d’ajouter des guides multiples par oligonucléotide, des homologies génomiques ou des promoteurs minimaux.
Regardez notre webinaire : Criblages CRISPR de nouvelle génération basés sur la transcriptomique en cellule unique
Banques de données génomiques relatives aux puces à sonde recouvrantes
Les banques de puces permettent une étude complète de la fonctionnalité génomique. Les pools d’oligonucléotides peuvent coder des jeux ordonnés de peptides, des séquences codant des ARNm ou des fragments de génome.
Les avantages du produit sont les suivants :
- Le « tiling » détermine les régions génomiques d’intérêt. Réduire le bruit expérimental en excluant les séquences indésirables à l’étape de la conception.
- Les séquences complexes, répétitives ou palindromiques qui ne peuvent généralement pas être synthétisées sous forme de gènes peuvent être codées dans des oligonucléotides.
- Les structures peptidiques plus importantes ou les architectures de génomes peuvent être codées dans des oligos de 300 nt de long.
- La synthèse uniforme minimise le suréchantillonnage nécessaire à un criblage complet.
Lisez notre article : Identification des facteurs de virulence du coronavirus pour le développement de vaccins à ARN avec la méthode fate-seq basée sur le pool d’oligonucléotides de Twist Bioscience.
Cribles de contrôle génétique à haut débit
La compréhension des éléments génétiques contrôlant l’expression et la régulation des gènes trouve une large application dans la biologie de synthèse et le développement de traitements potentiels des maladies. Les pool d’oligonucléotides permettent de réaliser des cribles à haut débit du contrôle génétique, notamment le profilage de l’expression génétique et les applications basées sur les rapporteurs.
Les avantages du produit sont les suivants :
- Des pools de haute qualité permettent de vérifier la fonctionnalité de chaque base.
- Une grande uniformité signifie que vous criblerez moins pour trouver vos réponses.
- Des pools de 300 nt vous donnent la possibilité d’ajouter plusieurs éléments ou régions d’homologie.
Regardez notre webinaire : Criblage épitranscriptomique sur TechNetworks
Hybridation in situ en fluorescence
L’hybridation in situ par fluorescence (FISH) permet de visualiser directement l’abondance et l’organisation spatiale des acides nucléiques dans les échantillons biologiques. Les pool d’oligonucléotides peuvent coder des sondes qui, lorsqu’elles sont liées à des marqueurs fluorescents, permettent une visualisation programmable et réglable d’un grand nombre de cibles.
Les avantages du produit sont les suivants :
- Développement rapide de sondes FISH
- Le contrôle de la séquence permet un ciblage efficace
- Des oligonucléotides plus longs permettent un ciblage plus spécifique
Regardez notre webinaire : Production rapide et efficace de sondes FISH à l’aide de pools d’oligonucléotides, avec Nicola Crosetto
Webinaire
Criblages CRISPR de nouvelle génération basés sur la transcriptomique en cellule unique
Webinaire
Utilisation de la composition et de la conservation des acides aminés pour des criblages CRISPR plus efficaces
Webinaire
Production rapide et efficace de sondes FISH à l’aide de pools d’oligonucléotides
Webinaire
Recherche des mécanismes de résistance de l’inhibiteur de PARP à l’aide de criblages CRISPR focalisés et à l’échelle du génome
Protocole
Ces directives décrivent les meilleures pratiques pour remettre en suspension les fragments de gènes Twist, les gènes clonaux, les pools d’oligonucléotides et les banques de variants.
Webinaire
Criblages CRISPR de nouvelle génération basés sur la transcriptomique en cellule unique
Webinaire
Utilisation de la composition et de la conservation des acides aminés pour des criblages CRISPR plus efficaces
Webinaire
Production rapide et efficace de sondes FISH à l’aide de pools d’oligonucléotides
Webinaire
Recherche des mécanismes de résistance de l’inhibiteur de PARP à l’aide de criblages CRISPR focalisés et à l’échelle du génome
Protocole
Ces directives décrivent les meilleures pratiques pour remettre en suspension les fragments de gènes Twist, les gènes clonaux, les pools d’oligonucléotides et les banques de variants.