La pandémie de Coronavirus a suscité une réponse rapide des équipes de recherche et de traitement du monde entier. Ces équipes de chercheurs ont besoin d’outils génomiques solides et fiables pour classifier et caractériser les échantillons de virus. Twist propose des panels d’enrichissement de cibles pour NGS « Uniquement à usage de recherche » (RUO) pour la détection et la caractérisation du virus SARS-CoV-2. Le panel cible environ 30 kb de génome viral avec environ 1 000 sondes, conçues contre le génome du SARS-CoV-2 (GenBank : MN908947.3).
Présentation de l’analyse NGS du SARS-CoV-2 - Le protocole RUO est une analyse hautement sensible d’hybridation d’acide nucléique basée sur capture et réservée à la recherche qui est utilisée pour la détection, la caractérisation et la surveillance environnementale du virus du SARS-CoV-2. Il s’appuie sur la capacité unique de Twist Bioscience à rapidement développer des panels spécifiques à des virus par synthèse d’ADN et sur le logiciel d’analyse exhaustive de données et les capacités de production de rapports (COVID-DX (v1.0)) de Biotia.
- Niveau de détection sensible de seulement 800 copies virales par ml dans chaque spécimen
- Couverture > 99,9 % du génome avec un post-enrichissement de 1X ou plus.
- Capacité d’identifier les nouvelles mutations virales
- Le rapport analytique du logiciel RUO fournit un typage des variants et une analyse phylogénétique pour la surveillance épidémiologique

La pandémie de Coronavirus a suscité une réponse rapide des équipes de recherche et de traitement du monde entier. Ces équipes de chercheurs ont besoin d’outils génomiques solides et fiables pour classifier et caractériser les échantillons de virus. Twist propose des panels d’enrichissement de cibles pour NGS « Uniquement à usage de recherche » (RUO) pour la détection et la caractérisation du virus SARS-CoV-2. Le panel cible environ 30 kb de génome viral avec environ 1 000 sondes, conçues contre le génome du SARS-CoV-2 (GenBank : MN908947.3).
Présentation de l’analyse NGS du SARS-CoV-2 - Le protocole RUO est une analyse hautement sensible d’hybridation d’acide nucléique basée sur capture et réservée à la recherche qui est utilisée pour la détection, la caractérisation et la surveillance environnementale du virus du SARS-CoV-2. Il s’appuie sur la capacité unique de Twist Bioscience à rapidement développer des panels spécifiques à des virus par synthèse d’ADN et sur le logiciel d’analyse exhaustive de données et les capacités de production de rapports (COVID-DX (v1.0)) de Biotia.
- Niveau de détection sensible de seulement 800 copies virales par ml dans chaque spécimen
- Couverture > 99,9 % du génome avec un post-enrichissement de 1X ou plus.
- Capacité d’identifier les nouvelles mutations virales
- Le rapport analytique du logiciel RUO fournit un typage des variants et une analyse phylogénétique pour la surveillance épidémiologique

NGS pour la détection des virus
Le séquençage de nouvelle génération (NGS) permet une identification spécifique et à haut débit des infections dans les échantillons, notamment le sang, les écouvillons nasaux, les matières fécales, etc. Dans le cas d’infections virales, cependant, le séquençage direct peut représenter un enjeu en raison de la concentration extrêmement faible des virus d’intérêt et de la présence d’un bruit de fond élevé provenant de l’hôte La capture de cible qui fait appel à des sondes d’hybridation basées sur l’ADN pour isoler des séquences spécifiques à partir d’un échantillon génomique mixte, peut augmenter la sensibilité et la spécificité des recherches basées sur le NGS.
La capture de cible hautement sensible et précise de Twist permet une détection efficace de l’ARN viral. Nous démontrons la capacité de l’enrichissement ciblé à capturer et à détecter le génome viral à des nombres variables de copies virales ajoutées à l’ARN de référence humain. À une charge virale élevée (environ 1 million de copies), le génome complet est récupéré même avec seulement 25 000 lectures*. Alors que pour un nombre de copies plus faible, la couverture de grandes parties du modèle viral est maintenue. Aucune lecture du contrôle négatif ne correspond à la matrice virale, même en utilisant 8 millions de lectures mappées, ce qui démontre que notre détection à 10 copies représente une véritable capture et non une contamination de fond. Pour en savoir plus, consultez la note de l’application.
*Avec COVID-Dx, 1 million de lectures sont nécessaires pour l’analyse.
Le panel de recherche Twist SARS-CoV-2 est fourni avec un protocole simple à suivre depuis les échantillons d’ARN purifiés jusqu’au séquençage avec des composants facilement disponibles.

NGS pour la détection des virus
Le séquençage de nouvelle génération (NGS) permet une identification spécifique et à haut débit des infections dans les échantillons, notamment le sang, les écouvillons nasaux, les matières fécales, etc. Dans le cas d’infections virales, cependant, le séquençage direct peut représenter un enjeu en raison de la concentration extrêmement faible des virus d’intérêt et de la présence d’un bruit de fond élevé provenant de l’hôte La capture de cible qui fait appel à des sondes d’hybridation basées sur l’ADN pour isoler des séquences spécifiques à partir d’un échantillon génomique mixte, peut augmenter la sensibilité et la spécificité des recherches basées sur le NGS.
La capture de cible hautement sensible et précise de Twist permet une détection efficace de l’ARN viral. Nous démontrons la capacité de l’enrichissement ciblé à capturer et à détecter le génome viral à des nombres variables de copies virales ajoutées à l’ARN de référence humain. À une charge virale élevée (environ 1 million de copies), le génome complet est récupéré même avec seulement 25 000 lectures*. Alors que pour un nombre de copies plus faible, la couverture de grandes parties du modèle viral est maintenue. Aucune lecture du contrôle négatif ne correspond à la matrice virale, même en utilisant 8 millions de lectures mappées, ce qui démontre que notre détection à 10 copies représente une véritable capture et non une contamination de fond. Pour en savoir plus, consultez la note de l’application.
*Avec COVID-Dx, 1 million de lectures sont nécessaires pour l’analyse.
Le panel de recherche Twist SARS-CoV-2 est fourni avec un protocole simple à suivre depuis les échantillons d’ARN purifiés jusqu’au séquençage avec des composants facilement disponibles.

Biotia Analysis
Le panel de recherche Twist SARS-CoV-2 a été associé à des réactifs et des analyses supplémentaires pour créer une solution de bout en bout, de la collecte des échantillons à l’analyse tertiaire, aboutissant à l’analyse NGS SARS-CoV-2.
Le logiciel Biotia COVID-DX (v1.0) permet d’obtenir un rapport axé sur la recherche qui comprend la séquence complète du virus SARS-CoV-2, ce qui permet de mieux comprendre les mutations, les variations génétiques et l’évolution du virus lors de sa transmission. Les fichiers FASTQ (résultats du séquençage) peuvent être générés dans les laboratoires et soumis à Biotia COVID-DX (v1.0), un logiciel basé sur le cloud, pour générer des rapports destinés à la recherche uniquement. L’achat de tout kit d’analyse NGS SARS-CoV-2 comprend des crédits pour l’analyse bio-informatique COVID-DX (v1.0) de chaque échantillon.
Pour recevoir des crédits d’analyse Biotia, il suffit de s’inscrire sur le portail des utilisateurs Biotia via www.biotia.io et d’entrer le numéro de commande unique qui vous a été envoyé par courriel après avoir passé votre commande. Ces crédits donnent accès à des analyses d’échantillons grâce au système de génération de rapports du logiciel.
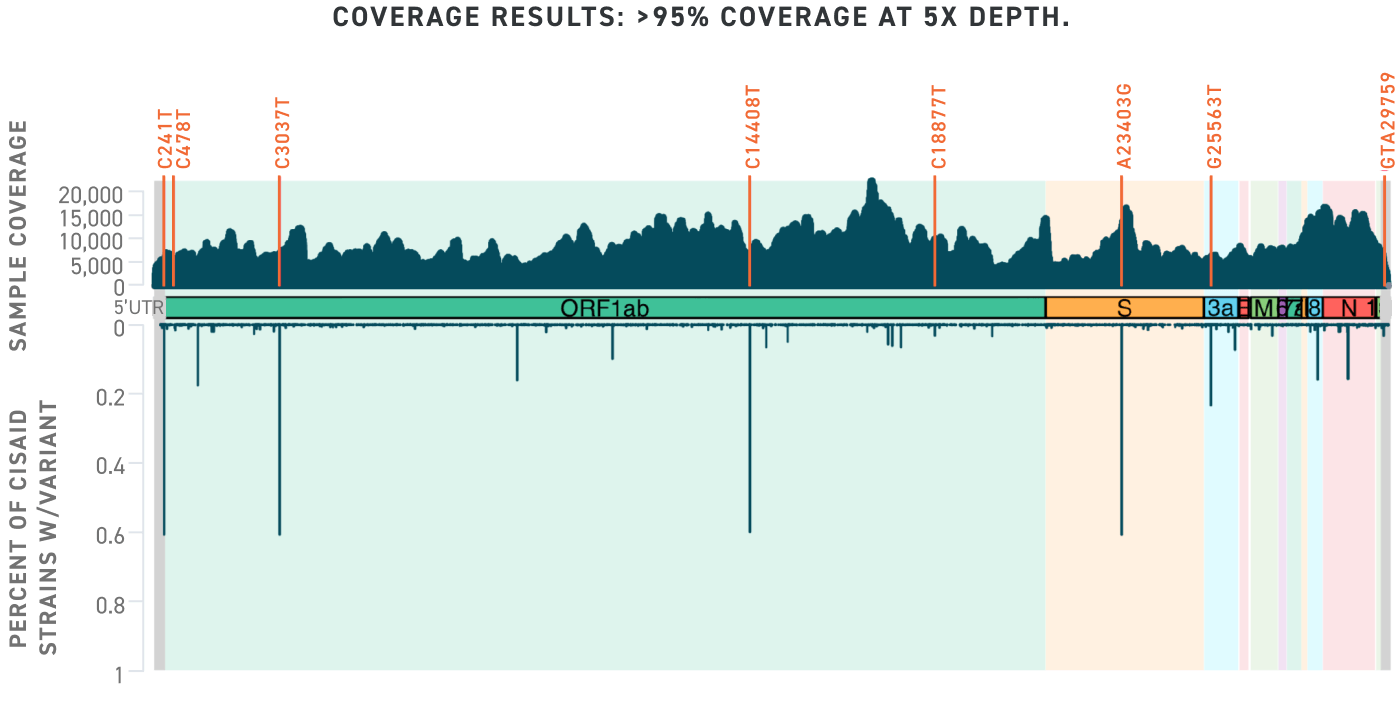
Les variants génétiques sont déterminés en comparant la souche testée au génome de référence du SARS-CoV-2 (NC_045512.2) et en indiquant l’emplacement/site du gène, l’altération et les notes explicatives, le cas échéant.
| SARS-CoV-2 détecté | Gène | Site (bp) | Altération | Valeur de référence |
| Oui | GORF1ab | 241 | T | Non détecté |
| Oui | ORF1ab | 478 | T | Non détecté |
| Oui | ORF1ab | 3 037 | T | Non détecté |
| Oui | ORF1ab | 14408* | T | Non détecté |
| Oui | ORF1ab | 18877 | T | Non détecté |
| Oui | S | 23403 | G | Non détecté |
| Oui | 3a | 25563 | T | Non détecté |
| Oui | 3’UTR | 29759 | C | Non détecté |
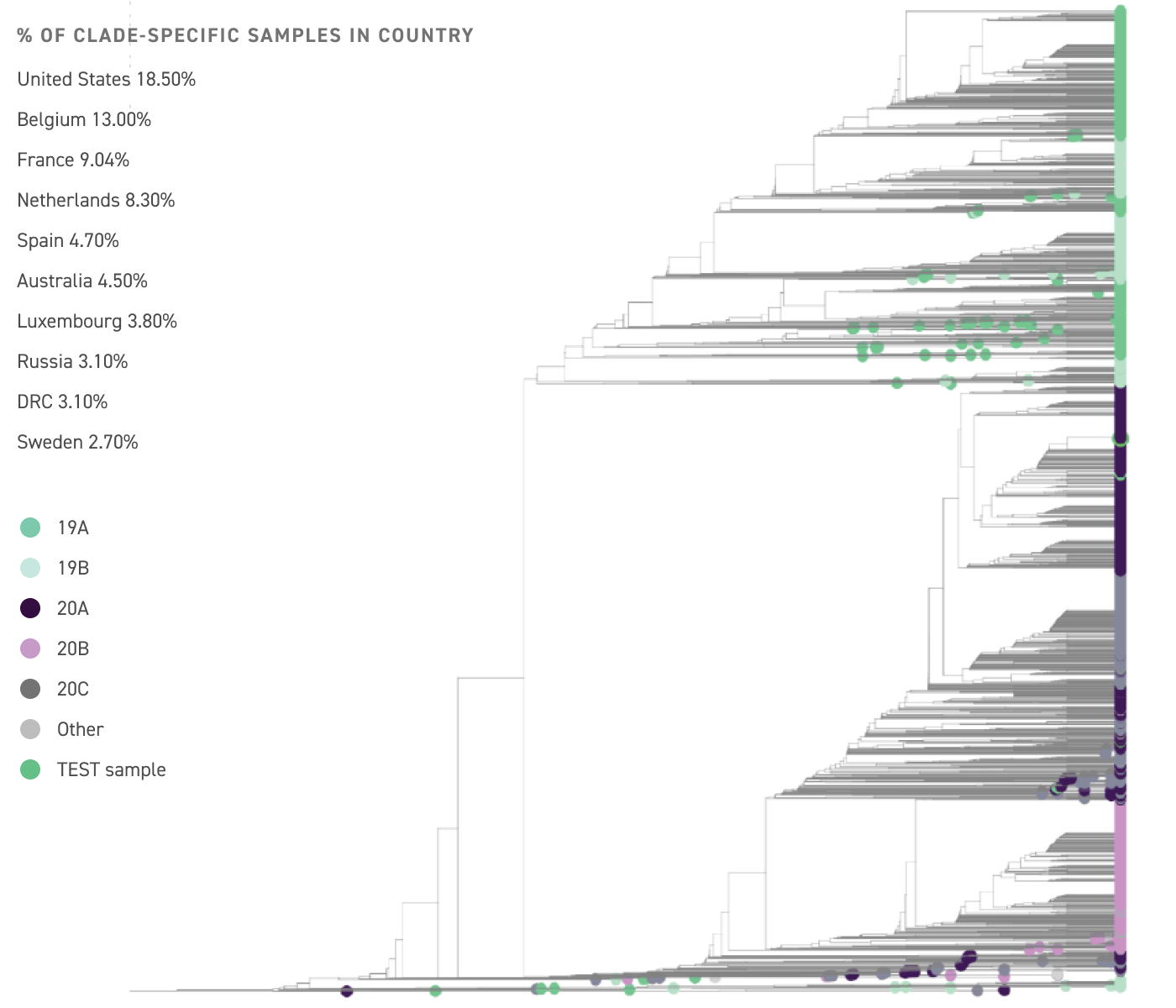
Le pourcentage du génome du SARS-CoV-2 récupéré dans votre échantillon testé et les variants génétiques identifiés par rapport au génome de référence sont indiqués. La proportion de variants génétiques connus des souches de SARS-CoV-2 provenant du monde entier est indiquée (en bas).
L’échantillon testé est plus étroitement lié au clade 20 A.
Ce clade est présent de manière très fréquente parmi les échantillons séquencés aux États-Unis, en Belgique et en France.
Biotia Analysis
Le panel de recherche Twist SARS-CoV-2 a été associé à des réactifs et des analyses supplémentaires pour créer une solution de bout en bout, de la collecte des échantillons à l’analyse tertiaire, aboutissant à l’analyse NGS SARS-CoV-2.
Le logiciel Biotia COVID-DX (v1.0) permet d’obtenir un rapport axé sur la recherche qui comprend la séquence complète du virus SARS-CoV-2, ce qui permet de mieux comprendre les mutations, les variations génétiques et l’évolution du virus lors de sa transmission. Les fichiers FASTQ (résultats du séquençage) peuvent être générés dans les laboratoires et soumis à Biotia COVID-DX (v1.0), un logiciel basé sur le cloud, pour générer des rapports destinés à la recherche uniquement. L’achat de tout kit d’analyse NGS SARS-CoV-2 comprend des crédits pour l’analyse bio-informatique COVID-DX (v1.0) de chaque échantillon.
Pour recevoir des crédits d’analyse Biotia, il suffit de s’inscrire sur le portail des utilisateurs Biotia via www.biotia.io et d’entrer le numéro de commande unique qui vous a été envoyé par courriel après avoir passé votre commande. Ces crédits donnent accès à des analyses d’échantillons grâce au système de génération de rapports du logiciel.
Les variants génétiques sont déterminés en comparant la souche testée au génome de référence du SARS-CoV-2 (NC_045512.2) et en indiquant l’emplacement/site du gène, l’altération et les notes explicatives, le cas échéant.
| SARS-CoV-2 détecté | Gène | Site (bp) | Altération | Valeur de référence |
| Oui | GORF1ab | 241 | T | Non détecté |
| Oui | ORF1ab | 478 | T | Non détecté |
| Oui | ORF1ab | 3 037 | T | Non détecté |
| Oui | ORF1ab | 14408* | T | Non détecté |
| Oui | ORF1ab | 18877 | T | Non détecté |
| Oui | S | 23403 | G | Non détecté |
| Oui | 3a | 25563 | T | Non détecté |
| Oui | 3’UTR | 29759 | C | Non détecté |
Le pourcentage du génome du SARS-CoV-2 récupéré dans votre échantillon testé et les variants génétiques identifiés par rapport au génome de référence sont indiqués. La proportion de variants génétiques connus des souches de SARS-CoV-2 provenant du monde entier est indiquée (en bas).

L’échantillon testé est plus étroitement lié au clade 20 A.
Ce clade est présent de manière très fréquente parmi les échantillons séquencés aux États-Unis, en Belgique et en France.

104796
Analyse NGS du SARS-CoV-2 Twist - RUO, 16 échantillons104797
Analyse NGS du SARS-CoV-2 Twist - RUO, 96 échantillons104799
Analyse NGS du SARS-CoV-2 Twist - RUO, 768 échantillons103564
Twist SARS-CoV-2 Panel de recherche + Biotia COVID DX (v1.0) - RUO, 2 réactions, Kit103566
Twist SARS-CoV-2 Panel de recherche + Biotia COVID DX (v1.0) - RUO, 12 réactions, Kit103567
Twist SARS-CoV-2 Panel de recherche + Biotia COVID DX (v1.0) - RUO, 96 réactions, Kit102016
Panel de recherche SARS-CoV-2 Twist, 2 réactions, Kit102017
Panel de recherche SARS-CoV-2 Twist, 12 réactions, Kit102018
Panel de recherche SARS-CoV-2 Twist, 96 réactions, KitLes contrôles synthétiques SARS-CoV-2 ARN de Twist sont destinés à la recherche uniquement et sont soumis à des restrictions d’utilisation supplémentaires, comme indiqué dans les Conditions d’approvisionnement de Twist.
104796
Analyse NGS du SARS-CoV-2 Twist - RUO, 16 échantillons104797
Analyse NGS du SARS-CoV-2 Twist - RUO, 96 échantillons104799
Analyse NGS du SARS-CoV-2 Twist - RUO, 768 échantillons103564
Twist SARS-CoV-2 Panel de recherche + Biotia COVID DX (v1.0) - RUO, 2 réactions, Kit103566
Twist SARS-CoV-2 Panel de recherche + Biotia COVID DX (v1.0) - RUO, 12 réactions, Kit103567
Twist SARS-CoV-2 Panel de recherche + Biotia COVID DX (v1.0) - RUO, 96 réactions, Kit102016
Panel de recherche SARS-CoV-2 Twist, 2 réactions, Kit102017
Panel de recherche SARS-CoV-2 Twist, 12 réactions, Kit102018
Panel de recherche SARS-CoV-2 Twist, 96 réactions, KitLes contrôles synthétiques SARS-CoV-2 ARN de Twist sont destinés à la recherche uniquement et sont soumis à des restrictions d’utilisation supplémentaires, comme indiqué dans les Conditions d’approvisionnement de Twist.
Note d’application
Capture de cible par NGS pour la détection et la caractérisation du SARS-CoV-2
Note d’application
Capture de cible par NGS pour la détection et la caractérisation du SARS-CoV-2
