Neue Virusspezies identifizieren
Das Auftreten der SARS-CoV-2-Pandemie zusammen mit dem Ausbruch der Affenpocken unterstreicht die Notwendigkeit verbesserter Instrumente zum Nachweis und zur Beobachtung neuartiger viraler Krankheitserreger.
Das Design dieses Panels beinhaltet die Verwendung des CATCH-Algorithmus (Compact Aggregation of Targets for Comprehensive Hybridization). Weitere Einzelheiten zu CATCH finden Sie unter Metsky, H.C. und Siddle, K.J. et al., „Capturing sequence diversity in metagenomes with comprehensive and scalable probe design“. Nature Biotechnology“, 37(2), 160–168 (2019). DOI: 10,1038/ s41587-018-0006-x
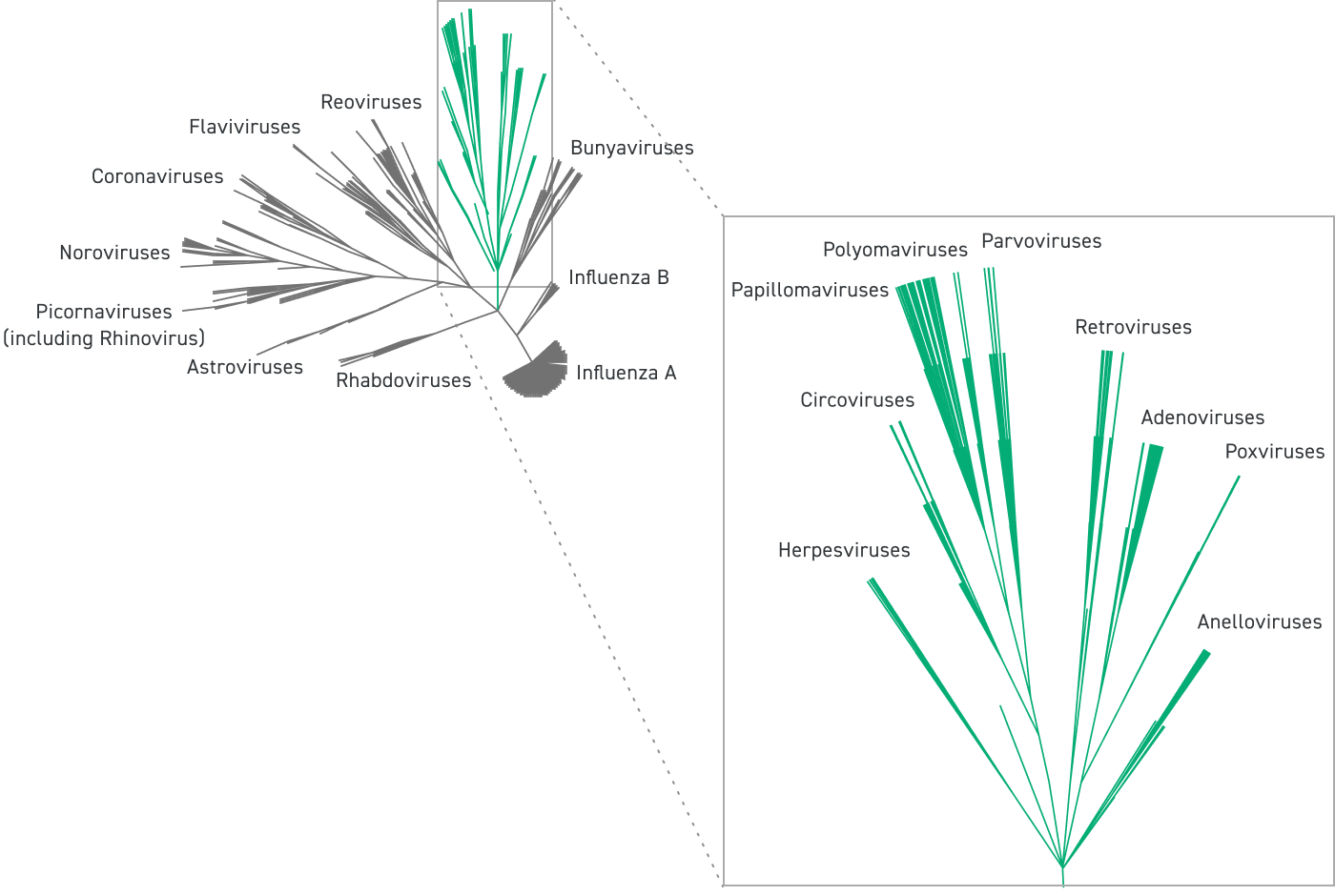
Das Twist Comprehensive Viral Research Panel behandelt Referenzsequenzen für 3.153 Viren, darunter 15.488 verschiedene Stämme. Beispiele für Viren, die identifiziert werden können:
Adenoviren
Coronaviren
Herpesviren
Influenza A und B
Affenpocken
Noroviren
Papillomaviren
Parvoviren
Picornaviren
Poxviren
Retroviren
Rhabdoviren
Alle Familien hier anzeigen
Neue Virusspezies identifizieren
Das Auftreten der SARS-CoV-2-Pandemie zusammen mit dem Ausbruch der Affenpocken unterstreicht die Notwendigkeit verbesserter Instrumente zum Nachweis und zur Beobachtung neuartiger viraler Krankheitserreger.
Das Twist Comprehensive Viral Research Panel behandelt Referenzsequenzen für 3.153 Viren, darunter 15.488 verschiedene Stämme. Beispiele für Viren, die identifiziert werden können:
Adenoviren
Coronaviren
Herpesviren
Influenza A und B
Affenpocken
Noroviren
Papillomaviren
Parvoviren
Picornaviren
Poxviren
Retroviren
Rhabdoviren
Alle Familien hier anzeigen
Das Design dieses Panels beinhaltet die Verwendung des CATCH-Algorithmus (Compact Aggregation of Targets for Comprehensive Hybridization). Weitere Einzelheiten zu CATCH finden Sie unter Metsky, H.C. und Siddle, K.J. et al., „Capturing sequence diversity in metagenomes with comprehensive and scalable probe design“. Nature Biotechnology“, 37(2), 160–168 (2019). DOI: 10,1038/ s41587-018-0006-x
Wir haben Referenzsequenzen aus den RefSeq-, FluDB- und VIPR-Datenbanken abgerufen, um die 1.052.421 einzigartigen Sonden zu entwickeln, aus denen das Twist Comprehensive Viral Research Panel besteht.
Das Panel umfasst im Großen und Ganzen jede Virusfamilie, die mindestens ein Virus enthält, von dem bekannt ist, dass es Menschen befällt, einschließlich Adenoviren, Coronaviren, Flaviviren, Noroviren, Influenzaviren und mehr.
Zusätzlich zu humaninfektiösen Viren umfasst das Panel auch tierische Viren (z. B. Fledermaus-Coronaviren). Dies ermöglicht den Nachweis neuer Virusarten unabhängig von ihrer Herkunft.
Aufgrund seines breiten Spektrums kann das Twist Comprehensive Viral Research Panel neuartige und weiterentwickelte Virusstämme nachweisen, einschließlich solcher, die im Zusammenhang mit einem Ausbruch der H1N1-Influenza bei Schweinefabrikarbeitern im Juni 2020 in den Mittelpunkt gerückt sind.
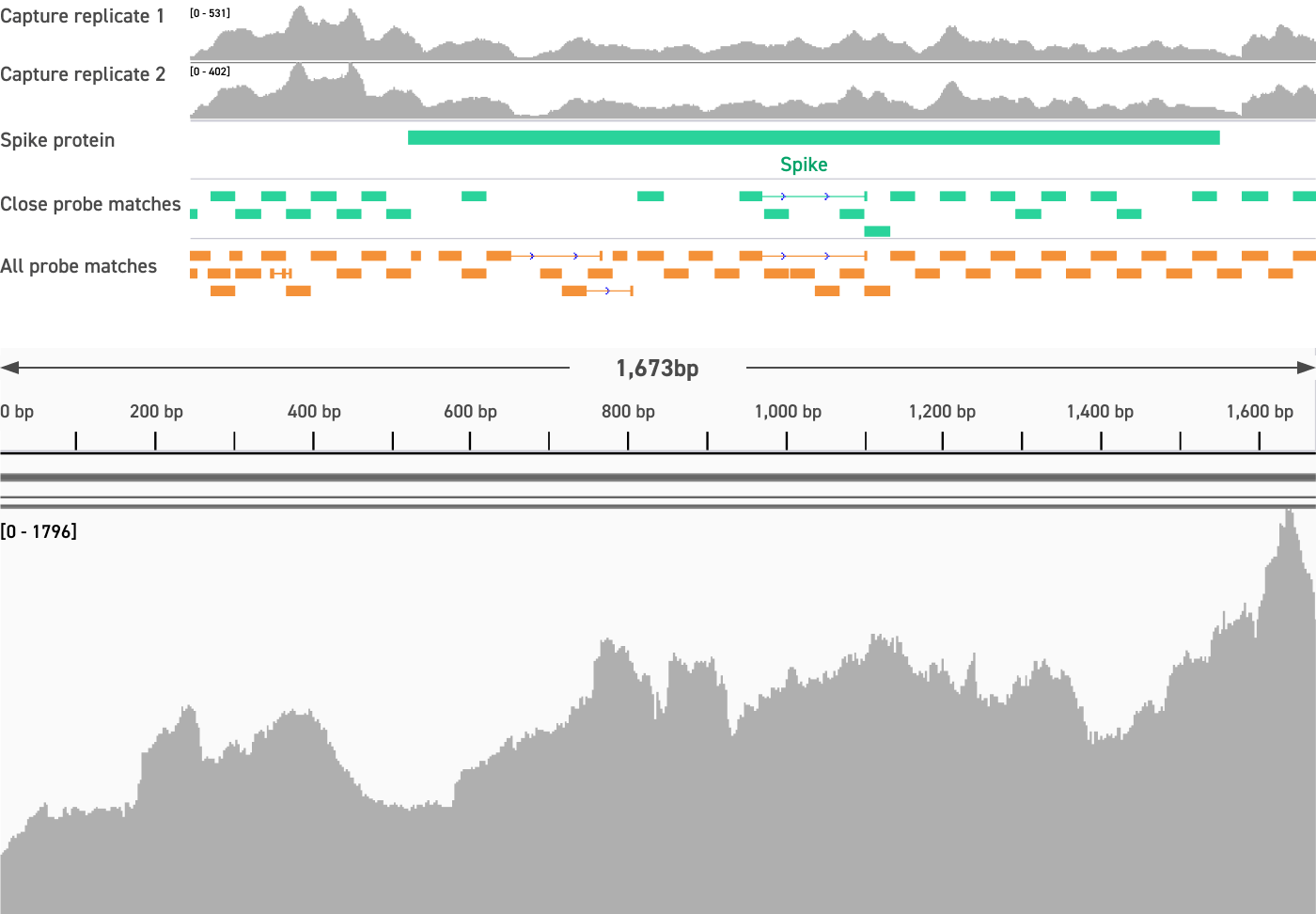
Die natürliche Selektion von Coronavirus-Spike- und Influenza-Hämagglutinin- (HA-) Glykoproteinen bei Tieren erleichterte die zoonotische Übertragung der SARS-CoV-2- und H1N1-Influenzaviren auf den Menschen.
Hier zeigen wir, dass das Twist Comprehensive Viral Research Panel verwendet werden kann, um diese schwer zu erfassenden Sequenzen erfolgreich zu erkennen, einschließlich der Spike-Region eines kürzlich nachgewiesenen Coronavirus (RoBat-CoV GCCDC1 Singapore) und eines HA-Segments, das bei der H1N1-Schweinegrippe im Juni 2020 isoliert wurde.
Unser Panel ergab eine Abdeckung jeder Sequenz von mehr als 99,8 % bis zu mindestens 1-facher Tiefe.
Wir definierten die Diskrepanzempfindlichkeit des Twist Comprehensive Viral Research Panel, indem wir eine Reihe von HA-Segmenten generierten, die definierte Niveaus der Sequenzvariation von 5 % bis 30 % von einem Wildtyp-HA-Segment enthielten.
Die vollständige Erfassung wurde bei 1-facher Abdeckung in Proben mit bis zu 10 % Sequenzvariation erreicht. Zusammen zeigen diese Daten, dass das Twist Comprehensive Viral Research Panel hochentwickelte Virussequenzen erfassen kann.
Der Multiplex-Nachweis von Viren kann mit dem Twist Comprehensive Viral Research Panel erfolgen, das metagenomische Anwendungen in einer Vielzahl von Probentypen ermöglicht.
In einem Koinfektionsassay konnte das Panel vier verschiedene Virustypen (Astrovirus – ssRNA, BK-Virus – dsDNA, Bocavirus – ssDNA und Picobirnavirus – dsRNA) mit nur 1,2 Millionen Lesezyklen erfolgreich in humane RNA einbringen.
Wir haben Referenzsequenzen aus den RefSeq-, FluDB- und VIPR-Datenbanken abgerufen, um die 1.052.421 einzigartigen Sonden zu entwickeln, aus denen das Twist Comprehensive Viral Research Panel besteht.
Das Panel umfasst im Großen und Ganzen jede Virusfamilie, die mindestens ein Virus enthält, von dem bekannt ist, dass es Menschen befällt, einschließlich Adenoviren, Coronaviren, Flaviviren, Noroviren, Influenzaviren und mehr.
Zusätzlich zu humaninfektiösen Viren umfasst das Panel auch tierische Viren (z. B. Fledermaus-Coronaviren). Dies ermöglicht den Nachweis neuer Virusarten unabhängig von ihrer Herkunft.
Aufgrund seines breiten Spektrums kann das Twist Comprehensive Viral Research Panel neuartige und weiterentwickelte Virusstämme nachweisen, einschließlich solcher, die im Zusammenhang mit einem Ausbruch der H1N1-Influenza bei Schweinefabrikarbeitern im Juni 2020 in den Mittelpunkt gerückt sind.

Die natürliche Selektion von Coronavirus-Spike- und Influenza-Hämagglutinin- (HA-) Glykoproteinen bei Tieren erleichterte die zoonotische Übertragung der SARS-CoV-2- und H1N1-Influenzaviren auf den Menschen.
Hier zeigen wir, dass das Twist Comprehensive Viral Research Panel verwendet werden kann, um diese schwer zu erfassenden Sequenzen erfolgreich zu erkennen, einschließlich der Spike-Region eines kürzlich nachgewiesenen Coronavirus (RoBat-CoV GCCDC1 Singapore) und eines HA-Segments, das bei der H1N1-Schweinegrippe im Juni 2020 isoliert wurde.
Unser Panel ergab eine Abdeckung jeder Sequenz von mehr als 99,8 % bis zu mindestens 1-facher Tiefe.

Wir definierten die Diskrepanzempfindlichkeit des Twist Comprehensive Viral Research Panel, indem wir eine Reihe von HA-Segmenten generierten, die definierte Niveaus der Sequenzvariation von 5 % bis 30 % von einem Wildtyp-HA-Segment enthielten.
Die vollständige Erfassung wurde bei 1-facher Abdeckung in Proben mit bis zu 10 % Sequenzvariation erreicht. Zusammen zeigen diese Daten, dass das Twist Comprehensive Viral Research Panel hochentwickelte Virussequenzen erfassen kann.
Der Multiplex-Nachweis von Viren kann mit dem Twist Comprehensive Viral Research Panel erfolgen, das metagenomische Anwendungen in einer Vielzahl von Probentypen ermöglicht.
In einem Koinfektionsassay konnte das Panel vier verschiedene Virustypen (Astrovirus – ssRNA, BK-Virus – dsDNA, Bocavirus – ssDNA und Picobirnavirus – dsRNA) mit nur 1,2 Millionen Lesezyklen erfolgreich in humane RNA einbringen.
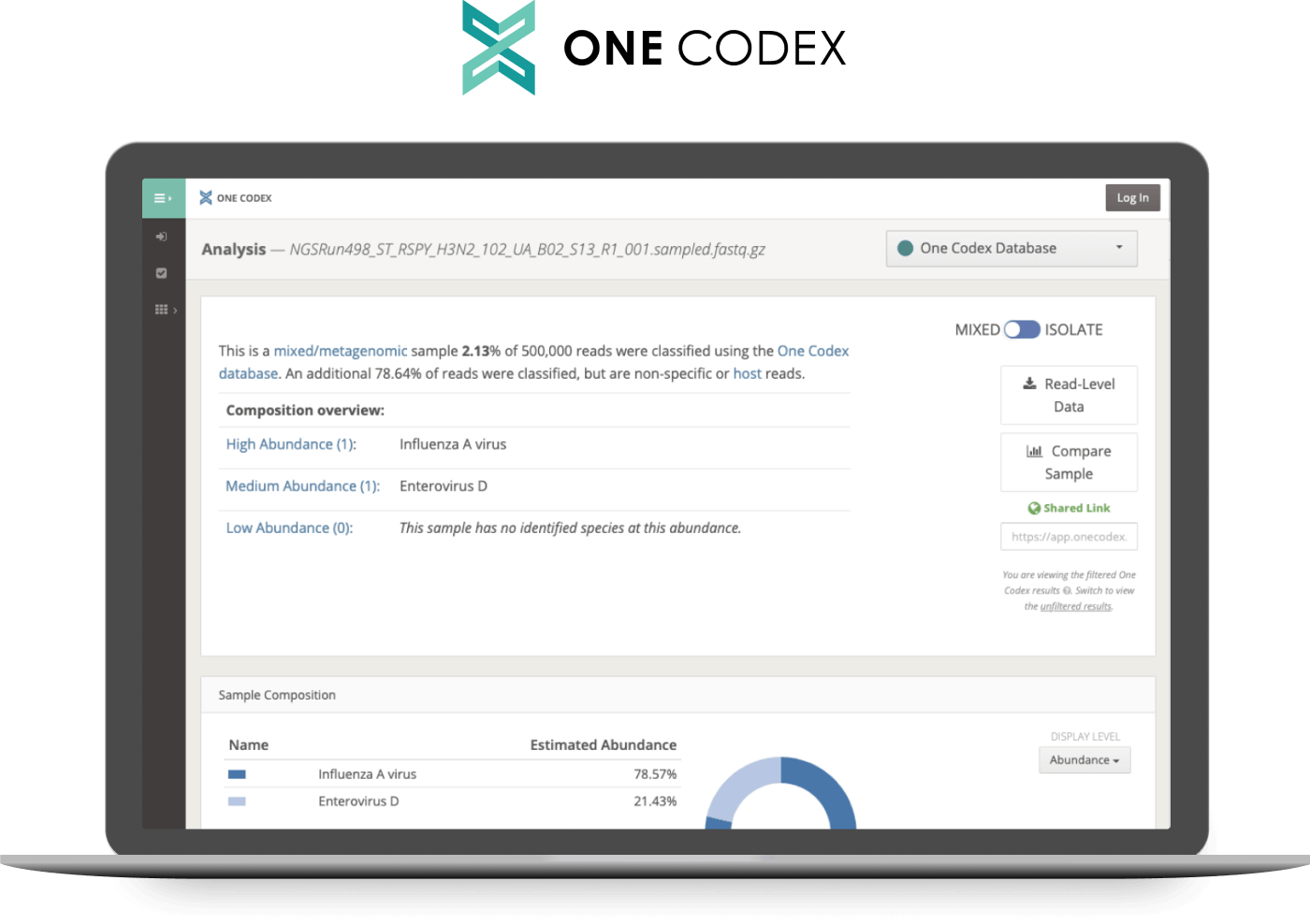
Das Twist Comprehensive Viral Research Panel bietet Zugriff auf das Webanalysetool One Codex. Dieses Cloud-basierte Tool bietet nicht nur Analyseunterstützung, sondern ermöglicht es Benutzern auch, ihre Bemühungen beim Nachweis neuartiger Viren mit druckbaren Berichten und veröffentlichungsbereiten Abbildungen zu dokumentieren.
Ein Codex vereinfacht die Analyse von Sequenzdaten, die mit dem Twist Comprehensive Viral Research Panel erhalten wurden. Nach dem Hochladen von Sequenzdaten mit einer einfachen Drag-and-Drop-Oberfläche können Benutzer innerhalb von Minuten einen Bericht erstellen, in dem die neuartigen Ergebnisse des Virennachweises aufgeführt sind. Benutzer können den Bioinformatik-Workflow auch mit ihren eigenen Erkennungsschwellen und -interpretationen anpassen.
Das Twist Comprehensive Viral Research Panel beinhaltet alles, was Sie für die Analyse Ihrer Ergebnisse mit One Codex benötigen, einschließlich eines Coupon-Codes und detaillierter Anweisungen. Weitere Informationen zu dieser optimierten Plattform für mikrobielle Analysen finden Sie unter onecodex.com
Das Twist Comprehensive Viral Research Panel bietet Zugriff auf das Webanalysetool One Codex. Dieses Cloud-basierte Tool bietet nicht nur Analyseunterstützung, sondern ermöglicht es Benutzern auch, ihre Bemühungen beim Nachweis neuartiger Viren mit druckbaren Berichten und veröffentlichungsbereiten Abbildungen zu dokumentieren.
Ein Codex vereinfacht die Analyse von Sequenzdaten, die mit dem Twist Comprehensive Viral Research Panel erhalten wurden. Nach dem Hochladen von Sequenzdaten mit einer einfachen Drag-and-Drop-Oberfläche können Benutzer innerhalb von Minuten einen Bericht erstellen, in dem die neuartigen Ergebnisse des Virennachweises aufgeführt sind. Benutzer können den Bioinformatik-Workflow auch mit ihren eigenen Erkennungsschwellen und -interpretationen anpassen.
Das Twist Comprehensive Viral Research Panel beinhaltet alles, was Sie für die Analyse Ihrer Ergebnisse mit One Codex benötigen, einschließlich eines Coupon-Codes und detaillierter Anweisungen. Weitere Informationen zu dieser optimierten Plattform für mikrobielle Analysen finden Sie unter onecodex.com

103545
Twist Comprehensive Viral Research Panel mit One Codex Software, 2 Reaktionen, Kit103547
Twist Comprehensive Viral Research Panel mit One Codex Software, 12 Reaktionen, Kit103548
Twist Comprehensive Viral Research Panel mit One Codex Software, 96 Reaktionen, KitWorkflow-Schritt
Target EnrichmentFormat
dsDNAPanelgröße
36,8 MbInhalt
VirussequenzenDesigndatenbanken
FluDB, RefSeq, VIPRdbLagerung
Bei -5 °C bis -30 °C lagern103545
Twist Comprehensive Viral Research Panel mit One Codex Software, 2 Reaktionen, Kit103547
Twist Comprehensive Viral Research Panel mit One Codex Software, 12 Reaktionen, Kit103548
Twist Comprehensive Viral Research Panel mit One Codex Software, 96 Reaktionen, KitWorkflow-Schritt
Target EnrichmentFormat
dsDNAPanelgröße
36,8 MbInhalt
VirussequenzenDesigndatenbanken
FluDB, RefSeq, VIPRdbLagerung
Bei -5 °C bis -30 °C lagern
Anwendungshinweis
Nachweis neuartiger Viren mit dem Twist Comprehensive Viral Research Panel
Anwendungshinweis
NGS-Zielanreicherung von viralen Krankheitserregern mit dem Twist Respiratory Virus Research Panel
Technisches Dokument
Liste der Twist Comprehensive Viral Research Panel-Stämme und -Spezies
Anwendungshinweis
Nachweis neuartiger Viren mit dem Twist Comprehensive Viral Research Panel
Anwendungshinweis
NGS-Zielanreicherung von viralen Krankheitserregern mit dem Twist Respiratory Virus Research Panel
Technisches Dokument
Liste der Twist Comprehensive Viral Research Panel-Stämme und -Spezies
