Las alternativas a la Cas9 permiten las aplicaciones CRISPR de última generación
Mejora del rendimiento de las cosechas. Eliminación de la Malaria en los mosquitos. Corrección de enfermedades genéticas anteriormente incurables. Siete años después del debut de la CRISPR-Cas9 como tijera genómica, la pregunta “¿qué puede hacer la Cas9?” se ha convertido en una pregunta difícil de responder. Después de todo, ¿qué es lo que no puede hacer la Cas9? Eliminemos su actividad nucleasa y la Cas9 se convierte en una plataforma modular para el control transcripcional, la modificación epigenética, el etiquetado genómico y la edición de bases, entre otras aplicaciones. El tremendo éxito biotecnológico de la Cas9 ha estimulado los esfuerzos por expandir la caja de herramientas CRISPR para incluir otras nucleasas CRISPR, lo que permitirá una nueva generación de aplicaciones CRISPR.
Las enzimas (Cas) asociadas a CRISPR existen en la mayoría de las arqueas y en muchas bacterias como un componente de su inmunidad adaptativa. En 2015, solo un par de años después de que la Cas9 se reconvirtiera a editora de genes, los sistemas Cas se organizaban en 2 clases, 5 tipos y 16 subtipos en función de sus características diferenciadoras. En la actualidad, el sistema de clasificación incluye 2 clases, 6 tipos y 33 subtipos (Makarova et al., 2020). Este cambio estuvo impulsado, en parte, por la búsqueda de nuevos sistemas de clase 2 que pudieran reconvertirse para la ingeniería genómica a la Cas9. Estos incluyen el editor de ADN Cas12a (Cpf1) y los editores de ARN basados en la Cas13.
Las argonautas, un grupo de proteínas guiadas por ácidos nucleicos mayoritariamente distintas de los sistemas CRISPR-Cas, recibieron mala prensa en 2016 cuando se retractó un artículo de perfil alto después de que varios grupos independientes no pudieran replicar sus supuestas capacidades de edición de genes (Gao et al., 2016). Sin embargo, las argonautas merecen una mención aquí, aunque solo sea para señalar una aplicación para la que son mejores que los sistemas CRISPR.
A continuación, se destacan algunos de los desarrollos más transformadores de la tecnología CRISPR (y de las argonautas) compilados por los expertos de Twist Bioscience.
Cas12 simplifica los cribados combinatorios
Muchos procesos biológicos, como las metástasis tumorales conllevan interacciones genéticas complejas que no pueden solucionarse por completo con un enfoque de bloqueo único. Aunque la Cas9 es el estándar de referencia para las aplicaciones de cribado de bloqueo único, su aplicación en cribados combinatorios se ha paralizado por sus capacidades subóptimas de multiplexación. Recientemente, varios grupos independientes han optimizado el sistema CRISPR de tipo V Cas12a (Cpf1) para proporcionar una alternativa mejorada a la Cas9 para la edición combinatoria de genes a escala (DeWeirdt et al., 2020; Gier et al., 2020).
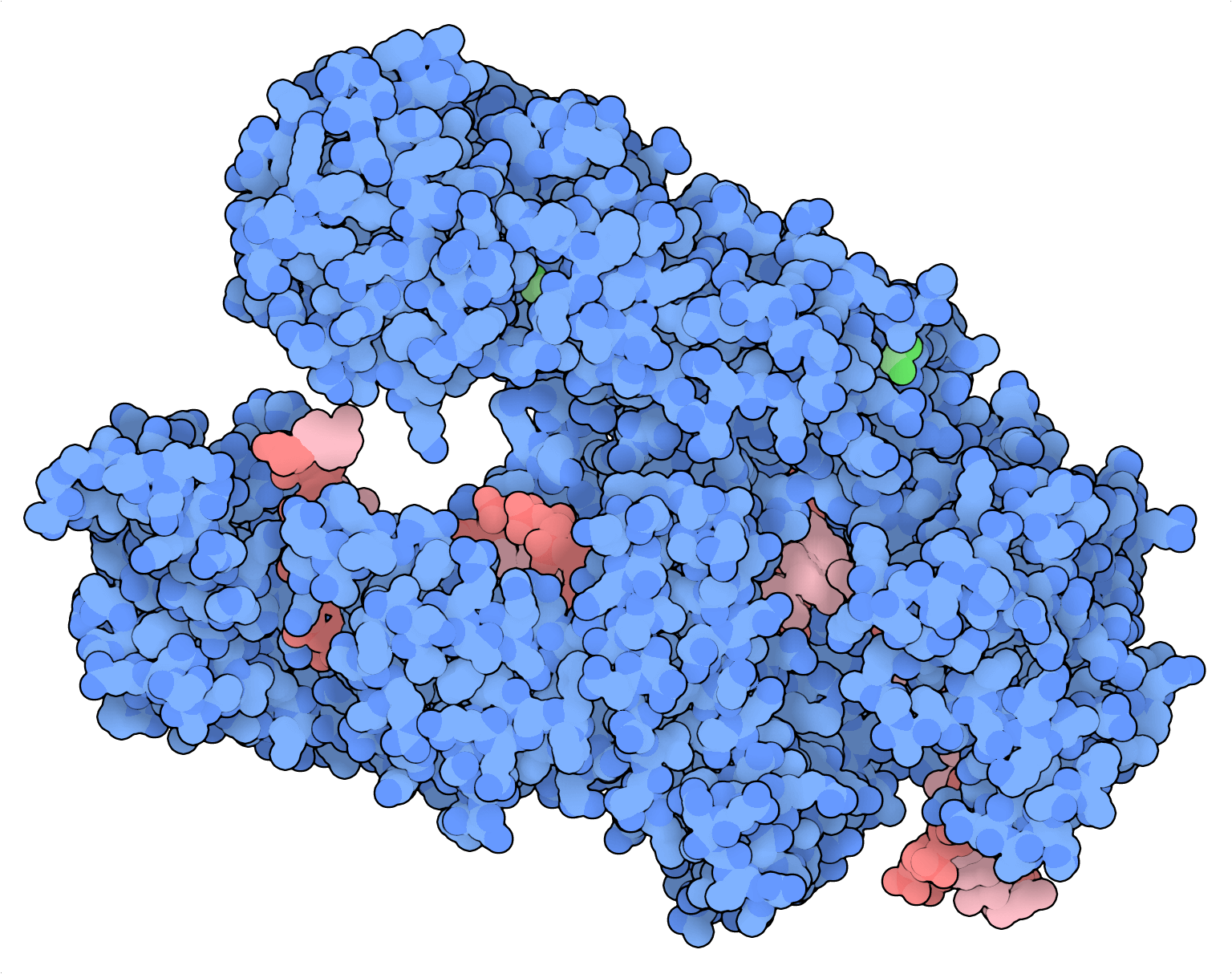
¿Qué hace de la Cas12a una opción mejor que la Cas9 para los cribados combinatorios? La Cas12a puede procesar matrices de ARN CRISPR (ARNcr) sin ARNasa III y ARN transactivador (ARNtracr), componentes necesarios para que la Cas9 cumpla la misma tarea. La Cas9 no puede procesar matrices de ARNcr por sí misma, por lo que las guías de Cas9 normalmente deben expresarse en casetes de expresión independientes, una por guía. Los elementos repetitivos en las guías de Cas9 multiplexadas provocan que tiendan a la recombinación y el desacoplamiento durante la administración de lentivirus, la PCR y la secuenciación profunda. Estos factores pueden complicar el diseño de bibliotecas, la clonación y el análisis.
Con Cas12a, los oligonucleótidos que contengan varios ARNcr pueden sintetizarse y clonarse con facilidad en vectores lentivirales para las aplicaciones de cribado combinatorio. Asimismo, los elementos repetitivos en los vectores de las guías de Cas12a multiplexadas son comparativamente más cortos y más tolerantes a las modificaciones en las secuencias, lo que hace que las guías de Cas12a multiplexadas sean menos vulnerables a la recombinación y el desacoplamiento durante el proceso de cribado (DeWeirdt et al., 2020; Gier et al., 2020). Un oligonucleótido de 300mer de Twist Bioscience puede codificar 3-4 guías de forma simultánea.
Aunque la Cas12a es una gran multiplexora, algunas de sus limitaciones incluyen una especificidad del motivo adyacente del protoespaciador (PAM) (5’-TTTV) más restrictiva y una eficiencia de mutación más baja que la Cas9. Estos parámetros se han mejorado mediante la ingeniería de proteínas guiada por estructuras de la Cas12a procedente de Acidaminococcus (AsCas12a; Kleinstiver et al., 2019) y han culminado en una enzima modificada denominada AsCas12a mejorada (enAsCas12a) que contiene varias mutaciones puntuales en residuos importantes para el reconocimiento del PAM. Además de expandir su especificidad con respecto al PAM (que ahora incluye TTYN, VTTV y TRTV, entre otros), se determinó de forma fortuita que la enAsCas12a mejoraba la eficiencia de edición al doble de media en comparación con la AsCas12a. La eficiencia del AsCas12a se ha mejorado todavía más añadiendo varias secuencias de localización nuclear y aclarando reglas de ARNcr fuera de objetivo y en objetivo (DeWeirdt et al., 2020; Gier et al., 2020).
Los sistemas CRISPR de tipo VI fijan sus miras en el ARN
El ARN de interferencia (ARNi) con ARN de horquilla corta (ARNhc) es una estrategia de knockdown de genes bien establecida que incorpora una vía de procesamiento de ARN endógeno para degradar ARNm celular. Sin embargo, el ARNi presenta efectos fuera de objetivo y una eficiencia incoherente del knockdown. El problema de especificidad de ha solucionado mediante la interferencia de CRISPR, un enfoque de knockdown que utiliza una Cas9 nucleasa muerta fusionada al dominio represor de la transcripción KRAB. Incluso así, algunas veces tanto la interferencia de CRISPR como la ARNi no aplican el knockdown de forma sólida sus objetivos en casos de una expresión génica basal alta o un mal procesamiento de las horquillas del ARNhc, respectivamente.
Introduzca RfxCas13d (CasRx), una nucleasa CRISPR dirigida al ARN y guiada por ARN. Puede que el CasRx no haya sido el primer editor de ARN descubierto, pero las comparaciones con otros sistemas Cas13 (LwaCas13a y PspCas13b) y los métodos existentes demuestran que el CasRx induce el knockdown más considerable en muchos genes, y con una alta especificidad de inicio. Y lo que es más, una dCasRx variante nucleasa muerta puede tener como objetivo los elementos cis del pre-ARNm para regular su empalme. Para destacar esta función, Konermann et al. (2018) establecieron como objetivo del dCasRx un silenciador de empalme intrónico para rectificar el empalme patológico del ARNm codificado por tau en un modelo neuronal de demencia frontotemporal.
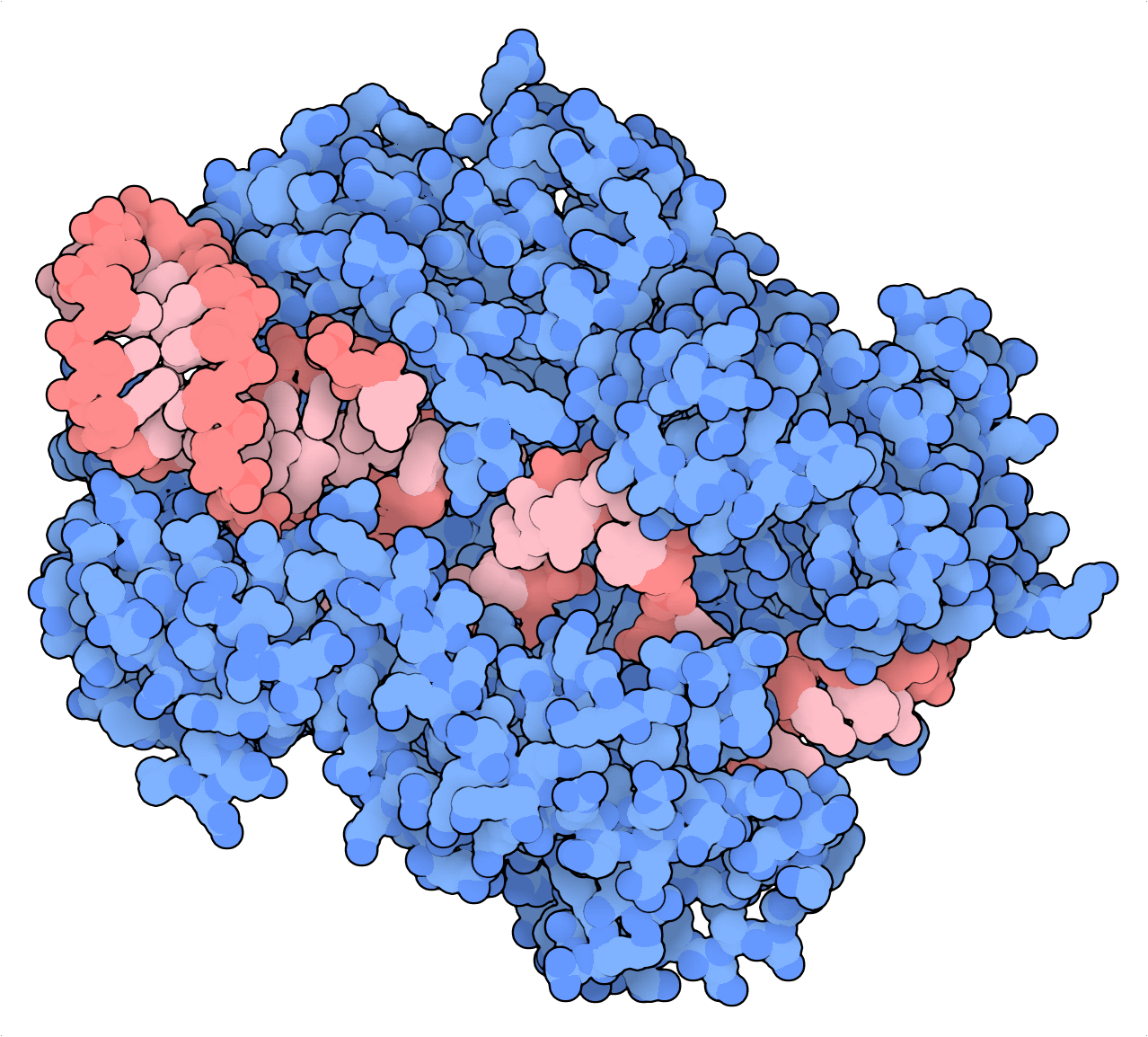
Los sistemas Cas13 tienen preferencia por el ARN monocatenario así que, en general, el direccionamiento de estos puede verse obstaculizado por una estructura secundaria de ARN (Abudayyeh et al., 2017). Sin embargo, su capacidad para provocar cambios transitorios al tener el ARN como objetivo en lugar del genoma y su falta de limitaciones en las secuencias les convierten en herramientas de edición de transcriptomas atractivas para aplicaciones experimentales y terapéuticas.
Los sistemas CRISPR/Cas de clase 1 diversifican el kit de herramientas CRISPR
A medida que los sistemas CRISPR/Cas de clase 2, como Cas9, Cas12s y Cas13s siguen dominando el espacio de la ingeniería del genoma, los sistemas CRISPR/Cas de clase 1 están empezando a atraer la atención por sus propiedades de escisión exclusivas y su composición multiproteína. Los más habituales entre estos son los sistemas de tipo I, que combinan la helicasa Cas3 con un complejo de proteínas Cas accesorias denominado complejo asociado a CRISPR para la defensa antiviral o “cascada”.
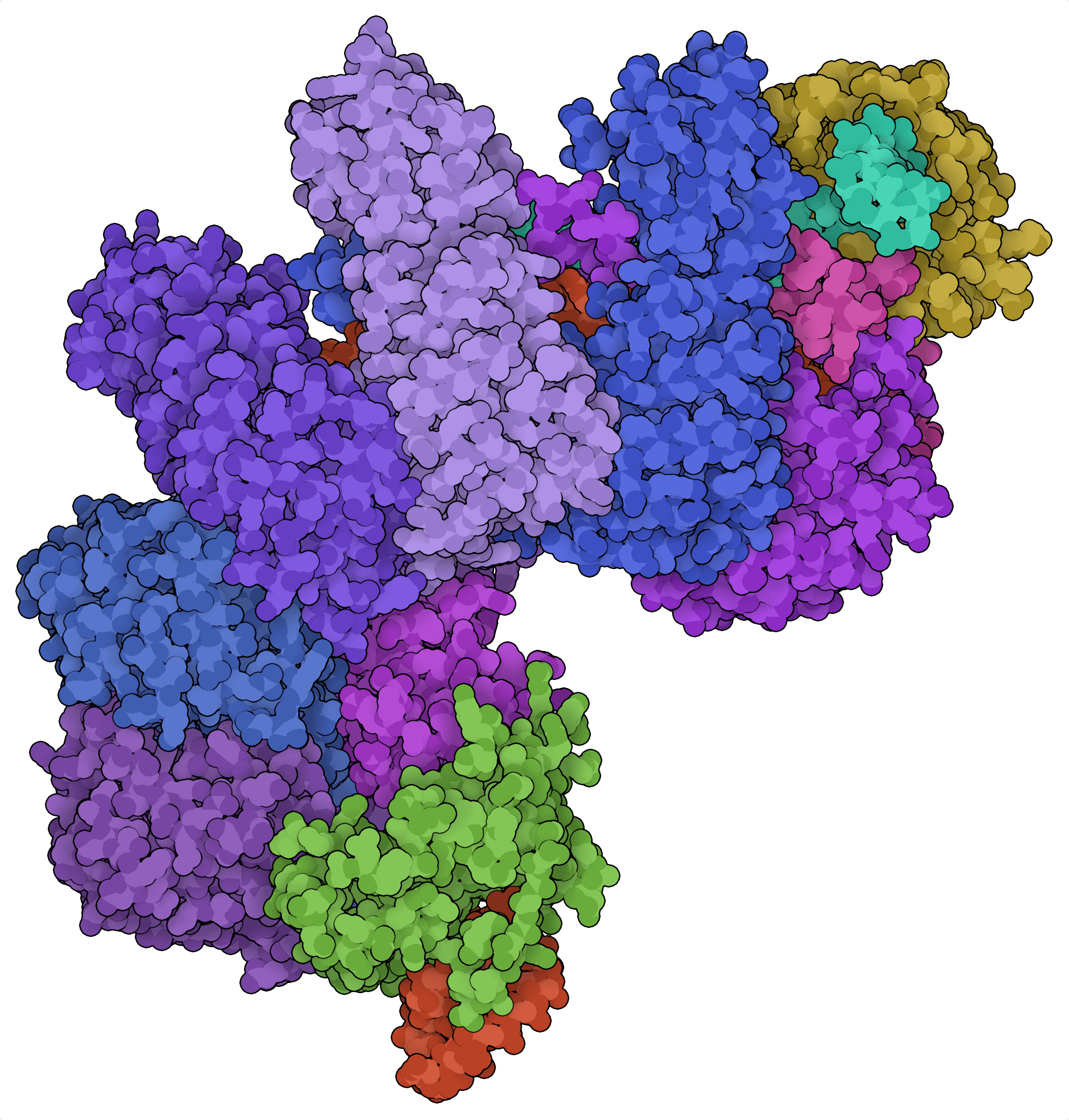
Si Cas9 y Cas12a son tijeras genómicas, Cas3 es una picadora genómica. En lugar de producir pequeñas indels como Cas9, Cas3 provoca diferentes eliminaciones genómicas de gran tamaño, que van de 0,5 a 100 kb de longitud, antes de los lugares genómicos objetivo (Dolan et al., 2019). Esta función promete nuevas aplicaciones de cribado de alto rendimiento en las que las bibliotecas de eliminación de mutantes pueden generarse con menos guías que los sistemas CRISPR de clase 2.
The Cascade complex alone also provides a modular platform for RNA-guided transcriptional activation and repression. Site-directed repression can be achieved using crRNAs in cas3-deleted E. coli (Luo et al., 2015; Rath et al., 2015) or in mammalian cells expressing exogenous Cascade proteins (Pickar-Oliver et al., 2019; Chen et al., 2020).
For gene activation, transcriptional activator domains need to be fused to one or more members of Cascade. Chen et al. (2020) obtained the best results when the synthetic VPR (VP64-p65-Rta) activator domain was fused to Csy3, the backbone component of Pseudomonas aeruginosa Cascade (PaeCascade) that interacts with the crRNA. Because multiple Csy3 proteins participate in the formation of the PaeCascade complex, multiple transcriptional effector domains can be recruited to a single Cascade complex. This strategy does not necessarily work for other class 1 systems, however, as the fusion of transcriptional effectors to Cas7 of EcoCascade (its Csy3 equivalent) failed to elevate transcription at targeted loci (Pickar-Oliver et al., 2019).
La naturaleza multiproteica de cascada resulta muy práctica para crear dispositivos modulares para el control transcripcional, pero también supone que las diferentes proteínas efectoras se coexpresen. Esto podría impedir el uso de sistemas de clase 1 para algunas aplicaciones.
Loci de CRISPR extraños revelan transposasas asociadas a CRISPR
Most CRISPR loci contain genes for site-directed nucleases. For example, CRISPR loci encoding type I systems typically contain cas3, which encodes the endonuclease effector of these Cas systems. Recent bioinformatic analyses, however, revealed CRISPR loci containing Tn7-like transposon genes instead of Cas endonuclease genes. From these loci, two CRISPR-associated transposon systems have been characterized: the class 2 type V-K effector Cas12k (Strecker et al., 2019) and the class 1 type I-F Cascade complex (Halpin-Healy et al., 2020). Instead of cleaving DNA, these Cas systems can insert large segments of DNA into the genome in a site-directed and nuclease-independent fashion.
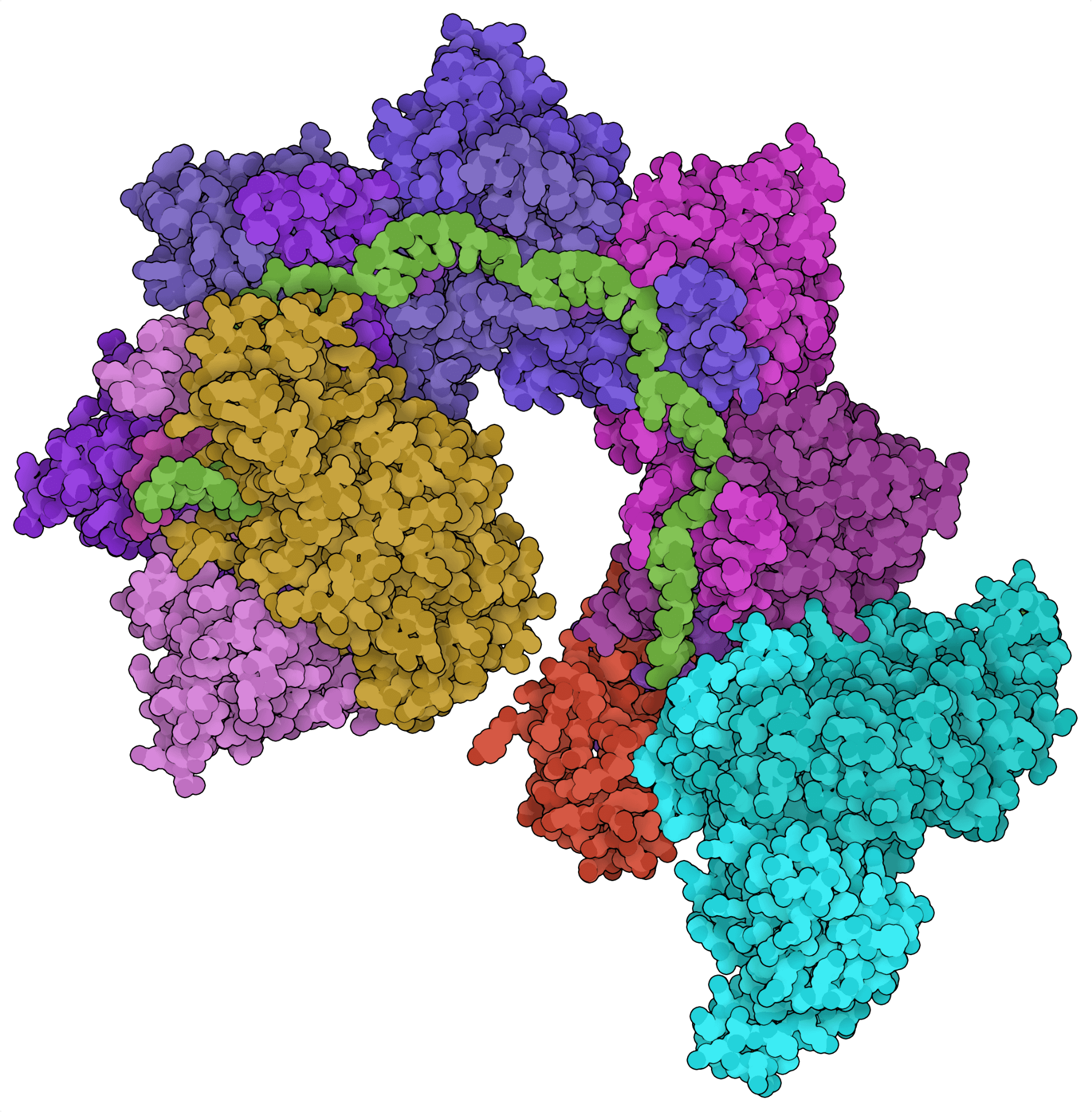
Los enfoques tradicionales de knock-in genético basado en CRISPR dependen de una reparación dirigida por homología tras un rotura de ADN dirigida al sitio específico: un proceso muy ineficaz. Los recientemente identificados transposones asociados a CRISPR ofrecen una solución alternativa potencialmente más eficiente, ya que permiten la transposición de fragmentos de ADN grandes. Las transposasas asociadas a CRISPR podrían utilizarse para sustituir exones defectuosos o generar linfocitos T con receptores de antígenos quiméricos (CAR). Aunque son prometedoras, todavía no se han demostrado las actividades de las transposasas de estos sistemas en las células eucariotas.
Las argonautas facilitan el almacenamiento de datos del ADN
Sometimes the best alternative to Cas9 is not a CRISPR system at all. This is true for nick-based DNA data storage, which requires a nicking enzyme to encode information in native DNA. DNA has been proposed as an alternative to current silicon-based storage media because of its relatively high durability and enormous storage density. To further push the limits of the information storage density of DNA, researchers have recently developed a nick-based approach (Tabatabaei et al., 2020).
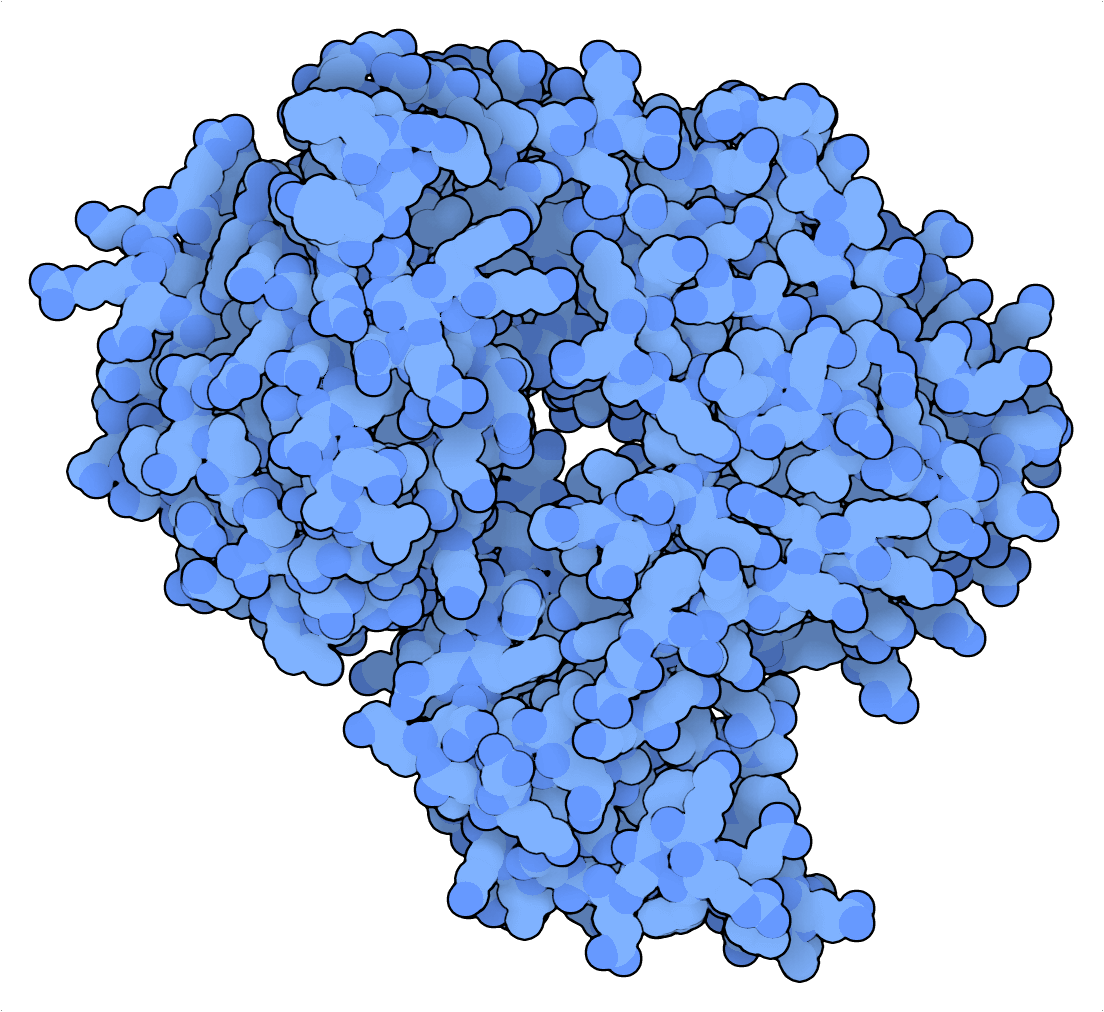
Tabatabaei et al. (2020) aprovecharon las propiedades favorables al mellado de PfArgo para crear un dispositivo de escritura en ADN que convierte una hebra de DNAbc en una “tarjeta perforada”. Aquí, la información binaria se codifica posicionalmente en un patrón de mellas, que se puede leer desnaturalizando fragmentos de una sola hebra de ADN. Posteriormente, estos fragmentos pueden secuenciarse mediante una secuenciación de última generación o por nanoporos, y asignarse a las tarjetas perforadas de referencia para recuperar la información codificada. Para resaltar la excepcional densidad de almacenamiento de esta estrategia, Tabatabaei et al. (2020) comprimieron un JPEG de 329 kilobytes (KB) del Lincoln Memorial en un archivo de ADN de 14 KB.
Para el almacenamiento de datos del ADN basado en mellas, las propiedades de mellado de la nickasa Cas9 palidece en comparación con las de la argonauta de Pyrococcus furiosus (PfAgo). Mientras que la primera es una enzima suicida que se basa en interacciones ARN-ADN inestables y restringidas por PAM para las dianas, la última puede inducir cientos de mellas por encima mediante guías de ADN cortas que no están limitadas por ninguna secuencia de dianas específica.
Estas son tan solo algunas de las aplicaciones posibles gracias a las proteínas guiadas por ácidos nucleicos en las CRISPR y las familias Argonauta. Para obtener más información sobre cómo expandir su caja de herramientas CRISPR, hable con un miembro del equipo.
Protein illustrations were generated from PDB structures using Illustrate, a non-photorealistic biomolecular illustration tool (Goodsell et al., 2019).
Referencias bibliográficas
Abudayyeh OO, Gootenberg JS, Essletzbichler P, Han S, Joung J, Belanto JJ, Verdine V, Cox DBT, Kellner MJ, Regev A, Lander ES, Voytas DF, Ting AY, Zhang F (2017) RNA targeting with CRISPR-Cas13. Nature 550:280-284.
Chen Y, Liu J, Zhi S, Zheng Q, Ma W, Huang J, Liu Y, Liu D, Liang P, Songyang Z (2020) Repurposing type I-F CRISPR-Cas system as a transcriptional activation tool in human cells. Nat Commun 11:3136.
DeWeirdt, PC, Sangree, AK, Hanna, RE et al. (2020) Genetic screens in isogenic mammalian cell lines without single cell cloning. Nat Commun 11, 752.
Dolan AE, Hou Z, Xiao Y, Gramelspacher MJ, Heo J, Howden SE, Freddolino PL, Ke A, Zhang Y (2019) Introducing a Spectrum of Long-Range Genomic Deletions in Human Embryonic Stem Cells Using Type I CRISPR-Cas. Mol Cell 74:936-950 e935.
Gao F, Shen XZ, Jiang F, Wu Y, Han C (2016) DNA-guided genome editing using the Natronobacterium gregoryi Argonaute. Nat Biotechnol 34:768-773.
Gier, RA, Budinich, KA, Evitt, NH et al. (2020) High-performance CRISPR-Cas12a genome editing for combinatorial genetic screening. Nat Commun 11, 3455
Goodsell, DS, Autin, L, Olson, AJ (2019) Illustrate: Software for Biomolecular Illustration. Structure 27:1716-1720.e1
Halpin-Healy TS, Klompe SE, Sternberg SH, Fernandez IS (2020) Structural basis of DNA targeting by a transposon-encoded CRISPR-Cas system. Nature 577:271-274.
Kleinstiver BP, Sousa AA, Walton RT, Tak YE, Hsu JY, Clement K, Welch MM, Horng JE, Malagon-Lopez J, Scarfo I, Maus MV, Pinello L, Aryee MJ, Joung JK (2019) Engineered CRISPR-Cas12a variants with increased activities and improved targeting ranges for gene, epigenetic and base editing. Nat Biotechnol 37:276-282.
Konermann S, Lotfy P, Brideau NJ, Oki J, Shokhirev MN, Hsu PD (2018) Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell 173:665-676 e614.
Luo ML, Mullis AS, Leenay RT, Beisel CL (2015) Repurposing endogenous type I CRISPR-Cas systems for programmable gene repression. Nucleic Acids Res 43:674-681.
Makarova KS et al. (2020) Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol 18:67-83.
Pickar-Oliver A, Black JB, Lewis MM, Mutchnick KJ, Klann TS, Gilcrest KA, Sitton MJ, Nelson CE, Barrera A, Bartelt LC, Reddy TE, Beisel CL, Barrangou R, Gersbach CA (2019) Targeted transcriptional modulation with type I CRISPR-Cas systems in human cells. Nat Biotechnol 37:1493-1501.
Rath D, Amlinger L, Hoekzema M, Devulapally PR, Lundgren M (2015) Efficient programmable gene silencing by Cascade. Nucleic Acids Res 43:237-246.
Strecker J, Ladha A, Gardner Z, Schmid-Burgk JL, Makarova KS, Koonin EV, Zhang F (2019) RNA-guided DNA insertion with CRISPR-associated transposases. Science 365:48-53.
Tabatabaei SK, Wang B, Athreya NBM, Enghiad B, Hernandez AG, Fields CJ, Leburton JP, Soloveichik D, Zhao H, Milenkovic O (2020) DNA punch cards for storing data on native DNA sequences via enzymatic nicking. Nat Commun 11:1742.
¿Qué piensa?
Me gusta
No me gusta
Me encanta
Me asombra
Me interesa
