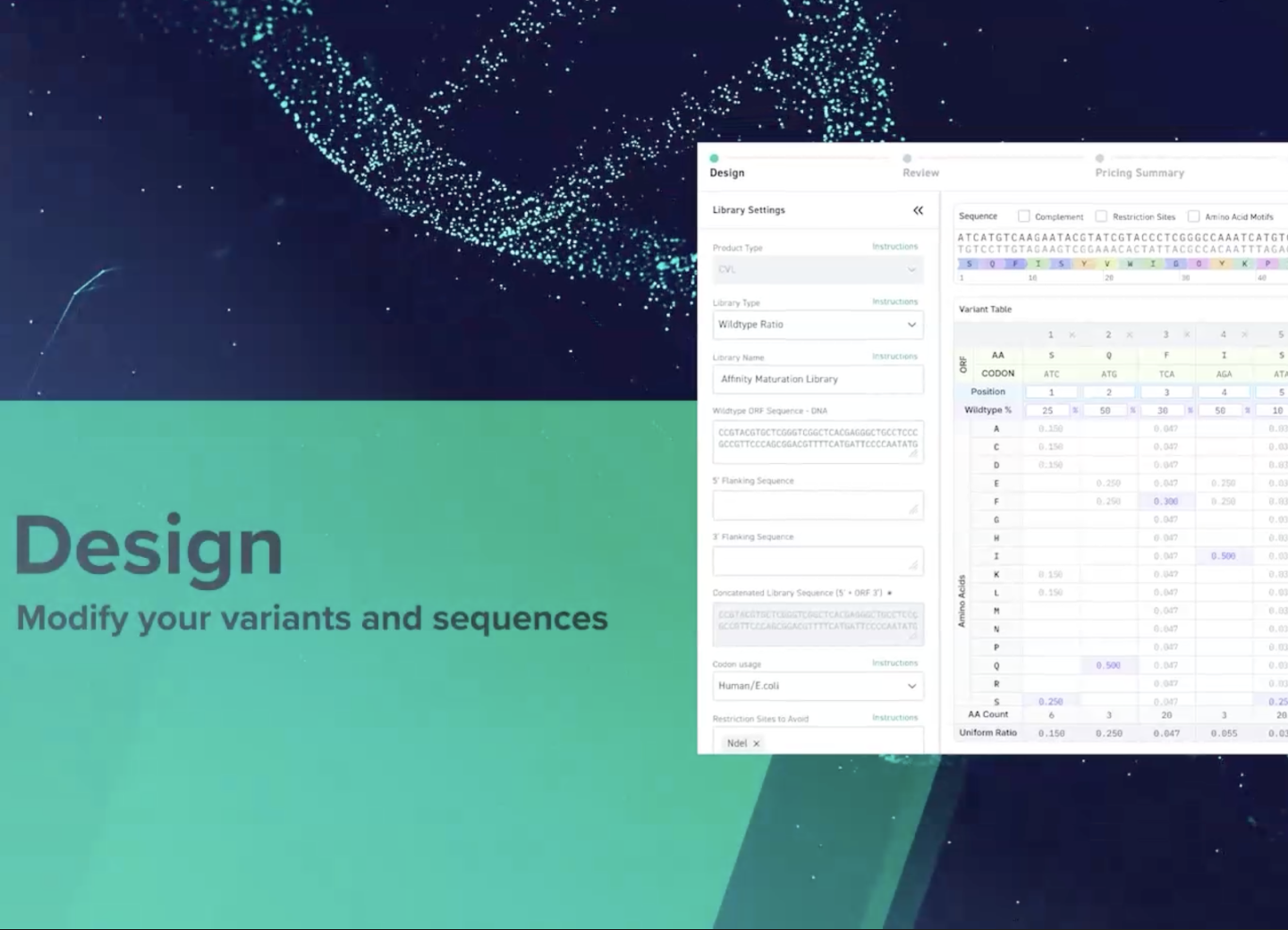
精密な変異体遺伝子ライブラリ合成技術で目的のスクリーニングを実現
Twist のシリコンベース超並列 DNA合成プラットフォームは、非常に均一かつ正確なオリゴヌクレオチドの製造を可能にします。90% のオリゴヌクレオチドが平均値の 2.5 倍未満で存在すると共に、業界トップクラスの低エラー率 1 : 2,000 塩基を実現しました。
Twist が持つ分子生物学の専門的基盤を活用したオリゴヌクレオチド合成プラットフォームにより、優れたバリアント表現と、不要なバイアスやモチーフのないユーザーの希望通りの組成を有する高多様性遺伝子変異体ライブラリの作製が可能です。Twist のライブラリテクノロジーは、変異体遺伝子の配列空間の網羅的な探索を可能にします。
限られた領域(例えば CDR)に複雑さや高多様性を有するライブラリを構築する Twist Bioscience の能力について、そして構築物の長さに多様性のある人工 DNA ライブラリの構築を可能にする技術の進歩について、詳しくご紹介します。
精密な変異体遺伝子ライブラリ合成技術で目的のスクリーニングを実現
Twist のシリコンベース超並列 DNA合成プラットフォームは、非常に均一かつ正確なオリゴヌクレオチドの製造を可能にします。90% のオリゴヌクレオチドが平均値の 2.5 倍未満で存在すると共に、業界トップクラスの低エラー率 1 : 2,000 塩基を実現しました。
Twist が持つ分子生物学の専門的基盤を活用したオリゴヌクレオチド合成プラットフォームにより、優れたバリアント表現と、不要なバイアスやモチーフのないユーザーの希望通りの組成を有する高多様性遺伝子変異体ライブラリの作製が可能です。Twist のライブラリテクノロジーは、変異体遺伝子の配列空間の網羅的な探索を可能にします。
限られた領域(例えば CDR)に複雑さや高多様性を有するライブラリを構築する Twist Bioscience の能力について、そして構築物の長さに多様性のある人工 DNA ライブラリの構築を可能にする技術の進歩について、詳しくご紹介します。
Twist Bioscience のシリコンベース DNA合成プラットフォームおよびライブラリテクノロジーにより、高信頼性のデータをより短期間で得られる高品質なライブラリがご利用いただけます。他の 2 つの競合技術と比較すると(図を参照)、 Twist のライブラリは設計したアミノ酸頻度からのずれが 1% 未満でした。Twist の in silico DNA合成プラットフォームによって、設計されたアミノ酸比率との正確な一致に加えて、望ましい結合モチーフや鎖長のバリエーションをマルチドメインライブラリにシームレスに組み込むことができるので、変異空間の網羅的解析を可能にする変異体遺伝子ライブラリを正確に設計し、カスタマイズすることができます。
また、 Twist ライブラリでは、NNK ライブラリや TRIM ライブラリにありがちな問題や課題を回避することができます。各変異体は塩基単位でプリントされ、合成前にスクリーニングされるため、終止コドン、悪影響を与えるモチーフ、不要な変異、望ましくないバイアスなどがすべてプロセスの最初に排除されます。その結果、機能面で要求通りの変異体がライブラリに濃縮され、スクリーニングにおける負荷が軽減されます。
業界をリードする当社のライブラリは、精密に設計された、すぐに使える高多様性ライブラリであり、より多くの機会で研究の目標を達成できます。
Twist には分子生物学の高度な専門的基盤があり、変異体遺伝子ライブラリを精密に構築することが可能です。当社の一塩基制御アプローチにより、スクリーニングプロセスに悪影響を与えるモチーフを排除した高多様性ライブラリをご提供可能です。当社は、目的とする変異をお客様自身が決めた比率で備えた、比類のない品質かつ完全カスタマイズのライブラリをご用意します。ここではその品質を表すコンビナトリアル変異ライブラリ (CVL) の例を示しています。7 つの連続したアミノ酸に関する変異体が作製され、それぞれの位置で予想通りの変異が見られ、ほぼすべてが目的とする比率になっています。
Position 1 と Position 6 では、野生型アミノ酸がそれぞれ 40%(Position 1)と 30%(Position 6)の比率になるよう設計されました。残りの 18 種のアミノ酸は、いずれも 3.3% 程度の低い割合となるよう設計されました。
Position 3 から Position 5 では、アミノ酸残基すべてが設計値、実測値とも 5.3%でした。
Twist Bioscience の精密な変異体遺伝子ライブラリでは、どのような CDR(相補性決定領域)配列をフレームワークに組み込むかを選択することができます。
各 CDR はコドン最適化が可能で、不要な制限部位が含まれないようにすることができます。機械学習はすでに、抗体ライブラリの解析や、例えばより高い親和性や特異性をもたらすような特定の CDR の組み合わせを同定するためのツールとして用いられており、科学研究に不可欠な要素となっています。
Twist のシリコンベース合成プラットフォームと組み合わせることで、解析から生成・明示されたライブラリの組み合わせを完全人工合成ライブラリにシームレスに組み込むことで、変異空間の探索を洗練させることができます。
当社のプラットフォームでは、高度に複雑なオリゴヌクレオチドを均一に合成することができ、下流のワークフローにおいて潜在的なバイアスを最小限に抑えることができます。当社の科学者チームは、ライブラリの作製とクローニングにおいて均一性を維持する方法を開発しました。一般的に、クローニングワークフローのステップはライブラリにバイアスをもたらしやすいことが知られており、スクリーニングやアッセイで一部の変異が不均衡に現れることにつながります。クローニングされたライブラリは、出発点の均一性を維持し、ドロップアウトや予定よりも変異導入が下回ってしまうことを最小限に抑えることで、高いレベルの多様性を維持することができ、スクリーニングの時間と労力の削減に貢献します。
この図は、作製後(Linear)とクローニング後(Cloned)の各変異位置で観察されたアミノ酸分布を、設計時の頻度分布(Expected)と比較したものです。
Twist Bioscience のシリコンベース DNA合成プラットフォームおよびライブラリテクノロジーにより、高信頼性のデータをより短期間で得られる高品質なライブラリがご利用いただけます。他の 2 つの競合技術と比較すると(図を参照)、 Twist のライブラリは設計したアミノ酸頻度からのずれが 1% 未満でした。Twist の in silico DNA合成プラットフォームによって、設計されたアミノ酸比率との正確な一致に加えて、望ましい結合モチーフや鎖長のバリエーションをマルチドメインライブラリにシームレスに組み込むことができるので、変異空間の網羅的解析を可能にする変異体遺伝子ライブラリを正確に設計し、カスタマイズすることができます。
また、 Twist ライブラリでは、NNK ライブラリや TRIM ライブラリにありがちな問題や課題を回避することができます。各変異体は塩基単位でプリントされ、合成前にスクリーニングされるため、終止コドン、悪影響を与えるモチーフ、不要な変異、望ましくないバイアスなどがすべてプロセスの最初に排除されます。その結果、機能面で要求通りの変異体がライブラリに濃縮され、スクリーニングにおける負荷が軽減されます。
業界をリードする当社のライブラリは、精密に設計された、すぐに使える高多様性ライブラリであり、より多くの機会で研究の目標を達成できます。
Twist には分子生物学の高度な専門的基盤があり、変異体遺伝子ライブラリを精密に構築することが可能です。当社の一塩基制御アプローチにより、スクリーニングプロセスに悪影響を与えるモチーフを排除した高多様性ライブラリをご提供可能です。当社は、目的とする変異をお客様自身が決めた比率で備えた、比類のない品質かつ完全カスタマイズのライブラリをご用意します。ここではその品質を表すコンビナトリアル変異ライブラリ (CVL) の例を示しています。7 つの連続したアミノ酸に関する変異体が作製され、それぞれの位置で予想通りの変異が見られ、ほぼすべてが目的とする比率になっています。
Position 1 と Position 6 では、野生型アミノ酸がそれぞれ 40%(Position 1)と 30%(Position 6)の比率になるよう設計されました。残りの 18 種のアミノ酸は、いずれも 3.3% 程度の低い割合となるよう設計されました。
Position 3 から Position 5 では、アミノ酸残基すべてが設計値、実測値とも 5.3%でした。
Twist Bioscience の精密な変異体遺伝子ライブラリでは、どのような CDR(相補性決定領域)配列をフレームワークに組み込むかを選択することができます。
各 CDR はコドン最適化が可能で、不要な制限部位が含まれないようにすることができます。機械学習はすでに、抗体ライブラリの解析や、例えばより高い親和性や特異性をもたらすような特定の CDR の組み合わせを同定するためのツールとして用いられており、科学研究に不可欠な要素となっています。
Twist のシリコンベース合成プラットフォームと組み合わせることで、解析から生成・明示されたライブラリの組み合わせを完全人工合成ライブラリにシームレスに組み込むことで、変異空間の探索を洗練させることができます。
当社のプラットフォームでは、高度に複雑なオリゴヌクレオチドを均一に合成することができ、下流のワークフローにおいて潜在的なバイアスを最小限に抑えることができます。当社の科学者チームは、ライブラリの作製とクローニングにおいて均一性を維持する方法を開発しました。一般的に、クローニングワークフローのステップはライブラリにバイアスをもたらしやすいことが知られており、スクリーニングやアッセイで一部の変異が不均衡に現れることにつながります。クローニングされたライブラリは、出発点の均一性を維持し、ドロップアウトや予定よりも変異導入が下回ってしまうことを最小限に抑えることで、高いレベルの多様性を維持することができ、スクリーニングの時間と労力の削減に貢献します。
この図は、作製後(Linear)とクローニング後(Cloned)の各変異位置で観察されたアミノ酸分布を、設計時の頻度分布(Expected)と比較したものです。
ご不明な点がございましたら、お気軽に {{[email protected]}} までメールでお問い合わせください
ご不明な点がございましたら、お気軽に {{[email protected]}} までメールでお問い合わせください